4 minute read
2. Metodología
mejora de protección de los cultivos, obteniendo como resultado un mejor rendimiento y calidad, lo que tiene un impacto positivo en la rentabilidad las explotaciones agrícolas de boniato.
2. Metodología
El material vegetal seleccionado en estos ensayos fueron tres clones de boniato: un clon del boniato tradicional valenciano tipo “Blanco”, y otros dos clones de carne anaranjada tipo “California” y “Málaga”. La selección se realizó en base a la demanda del mercado (productores y consumidores) y a la disponibilidad de variedades presentes en el banco de germoplasma del IVIA.
Se tomó muestra de las plantas madre y se realizó un primer muestreo para determinar los virus presentes en el material de partida. La idea principal fue recurrir a la puesta a punto de técnicas de PCR específicas, a nivel de especie o grupo, de cada uno de los virus descritos. En el método PCR se emplea un par de cebadores4 para reconocer zonas conservadas del genoma viral (ADN o ARN) de la muestra y definir la región de este material genético que será amplificada, en este caso, de los diferentes virus del boniato. Para ello se diseñaron cebadores de cada uno de los patógenos que venían descritos en la bibliografía como los virus que más afectaban al cultivo del boniato. Concretamente se ha desarrollado la metodología necesaria para detectar las especies virales SPCSV, SPLCV y las especies del género Potyvirus más importantes que afectan al boniato anteriormente mencionadas mediante diversas tecnologías basadas en PCR.
En primer lugar, se creó una colección de muestras positivas y negativas que se utilizaron como controles en las técnicas de PCR. Su manejo siempre se realizó de forma escrupulosa para minimizar así el riesgo de infecciones cruzadas. Los controles positivos y negativos se mantuvieron a -20 o -80ºC para evitar la pérdida de viabilidad o la degradación de su material genético.
La siguiente medida realizada, fue comprobar la especificidad de dichos cebadores ya que, a pesar de tratarse de cebadores descritos en artículos científicos, no siempre son específicos, y requieren ser revisados mediante estudios bioinformáticos y modificados adecuadamente para asegurarse una alta especificidad con cada uno de los virus estudiados (Figura 2). Por tanto, se verificó el correcto funcionamiento de los cebadores diseñados y se ensayaron diferentes concentraciones de cebadores para determinar los programas de PCR que presentaron mejores resultados en cuanto a repetitividad y reproducibilidad.
Además, paralelamente se iniciaron las labores de saneamiento del material vegetal de boniato mediante cultivo in vitro. Las metodologías de eliminación completa o ¨curación¨ de todos los patógenos presentes en una planta (saneamiento vegetal in vitro) se fundamenta en el cultivo de meristemos apicales, desde el aislamiento efectivo del ápice hasta el desarrollo de una planta completa enraizada. Para ello, mediante un microscopio binocular se procedió al aislamiento de
4 Cebador: Un cebador (también denominado iniciador, oligonucleótido o primer) es una secuencia corta de ADN de cadena simple que se utiliza en la reacción en cadena de la polimerasa (PCR). En el método PCR se emplea un par de cebadores para hibridar con el ADN diana (el que se quiere amplificar para detectar la presencia del ADN que se está analizando) y definir la región que será amplificada. Normalmente se necesitan dos cebadores, para indicar el inicio y el fin de la amplificación
ápices caulinares (aproximadamente entre 0,2-0,4 mm) en una cabina de flujo laminar, y cultivándolos en el medio apropiado hasta la obtención de la planta completa (Figura 3). Estas plantas procedentes del cultivo in vitro se aclimataron y se mantuvieron en el invernadero de cuarentena. Se tomaron muestras para garantizar el estado sanitario de las mismas, y una vez comprobado, se procedió a la multiplicación vegetativa de los tres clones seleccionados.
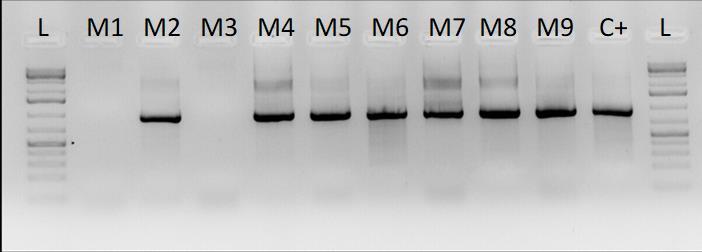
Figura 2. Detalle de electroforesis en gel de agarosa de una PCR convencional en búsqueda de muestras infectadas con Sweetpotato leaf curl virus. De izquierda a derecha se observa L (marcador de pesos moleculares), M1-M9 (muestras del campo de ensayos) y C+ (control positivo). En este caso, M1 y M3 resultaron ser las únicas muestras negativas para el virus analizado
Figura 3. Esquema del procedimiento del cultivo de meristemos de boniato para la obtención de planta sana

Una vez conseguidas las suficientes plantas saneadas, se realizó un ensayo agronómico empleando, de cada uno de los tres clones, plantas madre sin sanear y plantas saneadas por ValGenetics. El ensayo de campo se distribuyó con bloques al azar, con 10 plantas por bloque y tres repeticiones (total=30 plantas/clon/tratamiento), evaluando así un total de 180 plantas de boniato (Figura 4).
Se tomaron en campo distintas muestras de cada uno de los clones saneados y no saneados para confirmar la efectividad de las técnicas de diagnóstico puestas a punto. Durante el proceso de validación, se comprobó la especificidad de los cebadores y sondas mediante la secuenciación de los productos de PCR obtenidos, asegurando así la ausencia de contaminación cruzada debida a la posible presencia de múltiples microorganismos en las muestras de campo.
Por otro lado, se realizó una evaluación agronómica teniendo en cuenta la producción comercial de cada uno de los clones, y comparando los valores obtenidos con las plantas saneadas frente a las no saneadas.




