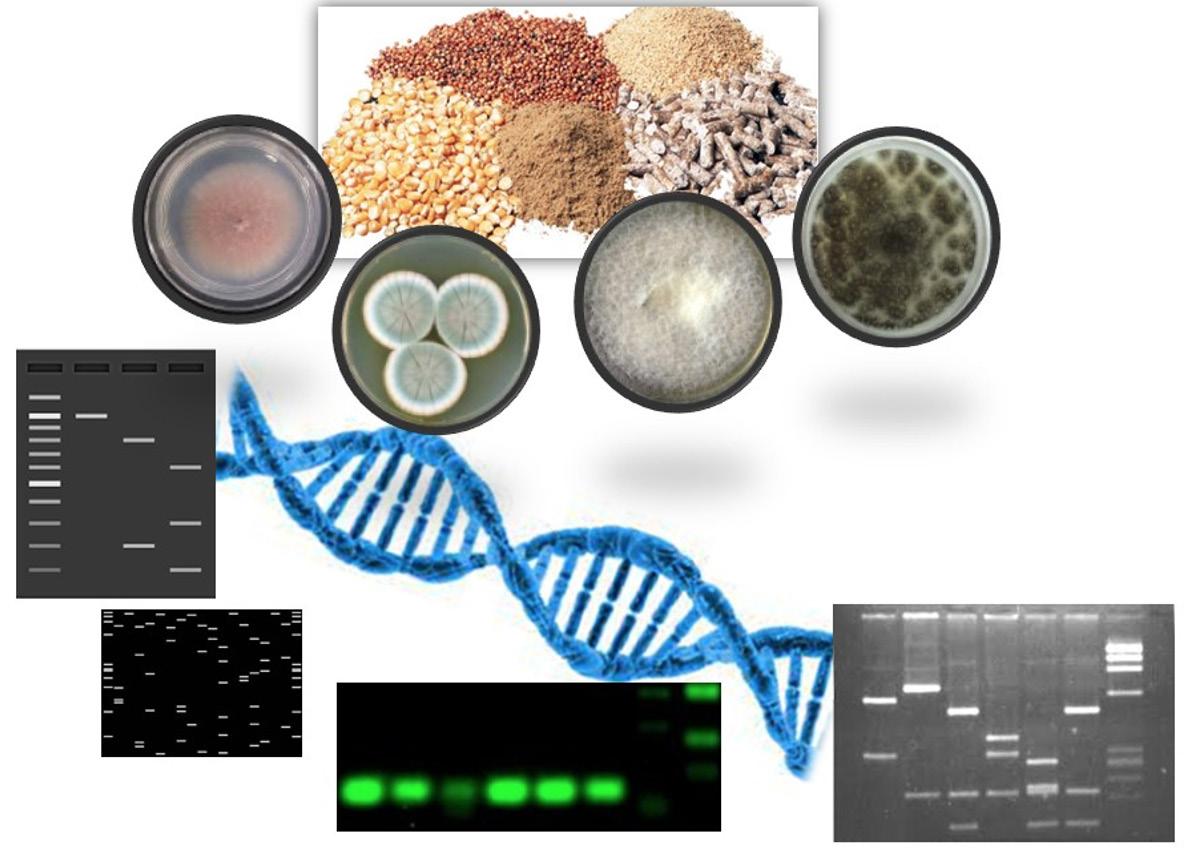
Diferentes técnicas moleculares utilizadas para la detección y diferenciación de hongos micotoxigénicos
PCR

La técnica PCR se utiliza para detectar la contaminación con hongos micotoxigénicos en matrices de piensos y para definir el riesgo potencial para la salud humana y animal
Esta técnica se emplea principalmente para la identificación de hongos y la detección de genes específicos.
Fusarium poae productor de nivalenol (NIV) se detectó mediante un ensayo de PCR que representa la presencia del gen tri7 (Dinolfo et al., 2012).
La implicación de la contaminación por F. verticillioides en 51 matrices de piensos se confirmó mediante PCR convencional y 69 muestras se asociaron con especies de Fusarium (Deepa et al., 2016a).
Las muestras de cereales para piensos estaban infectadas con 103 aislados de Fusarium y 64 F. verticillioides productores de fumonisina estaban asociados con el sorgo (Sreenivasa et al., 2007; 2008).
Entre 108 muestras de piensos, el 45,37% de las mezclas de piensos estaban contaminadas con especies de Fusarium, lo que se confirmó mediante un método de PCR convencional (Deepthi et al., 2017).
Dass et al. (2009) analizaron 80 mezclas de piensos para aves de corral y 114 piensos para animales en busca de contaminación por Fusarium, informando de 374 especies de Fusarium con cebadores y 244 aislados para cepas específicas de fumonisina con el conjunto de cebadores FUM1.
Implica una única ronda de PCR con un determinado conjunto de primers, reactivos de PCR y la muestra de ADN, seguida de un sistema de documentación en gel.
Ventajas Desventajas
El método es preciso y ofrece resultados rápidos.
Requiere mucha mano de obra y tecnología eficiente.
10
Reacción en cadena de la polimerasa (PCR) y sus variantes
1Kb
PCR Convencional
METODOLOGÍA
PCR MULTIPLEX

En un solo tubo de PCR, permite la combinación de varios cebadores específicos de la especie utilizados simultáneamente para detectar una variedad de hongos micotoxigénicos. Los productos amplificados se analizan mediante electroforesis en gel.
APLICACIÓN
En Brasil se analizaron muestras de maíz asociadas a la contaminación por F. verticillioides y F. subglutinans mediante el desarrollo del gen gaoB que codifica la galactosa oxidasa (Faria et al., 2012).
Se utilizaron un cebador directo y dos inversos para detectar F. verticillioides productor de fumonisina mediante mPCR (Deepa et al., 2016c).
Se detectaron especies de Aspergillus productoras y no productoras de aflatoxinas en meju, utilizándose dos conjuntos de cebadores para amplificar los genes (omtB, omtA, aflR, ver-1), conjunto I (omtB-1,2, aflR-1,2 y omtA-1,2) y conjunto II (aflR-1,2, ver-1-F-R y omtA-1,2) (Kim et al., 2011). Simultáneamente, cinco genes principales, pks (ocratoxina A), pks13 (zearalenona), aflr (aflatoxina), tri5 (tricoteceno) y fum13 (fumonisina) producidos por Aspergillus, Fusarium y Penicillium fueron detectados utilizando un ensayo de PCR multiplex para matrices de piensos (Priyanka et al., 2015).
Cuatro genes importantes relacionados con las micotoxinas, fum13 (fumonisina), nor1 (aflatoxina), Tri6 (tricoteceno) y otanps (ocratoxina A)se detectaron simultáneamente en una sola prueba mediante un método de PCR múltiplex específico y sensible (Rashmi et al., 2013).
Se detectaron las fumonisinas (FUM1, FUM13), y los tricotecenos (tri5, tri6) en matrices de piensos utilizando mPCR (Ramana et al., 2011).
Con esta técnica, una sola corrida de PCR es suficiente para la identificación de más de un gen a la vez.
PCR Multiplex 1Kb 400bp
Ventajas Desventajas
El método es preciso, rentable y ahorra tiempo.
Sensible
11
METODOLOGÍA APLICACIÓN
PCR ANIDADA
Este método implica dos reacciones consecutivas en un solo tubo cerrado, en el que el par de cebadores externos amplifica un gran amplicón utilizado como plantilla para el cebador interno que requiere una temperatura más alta que el cebador externo y donde el cebador interno no se unirá (Porter-Jordan et al., 1990).
El método es rentable y 100 veces más sensible que otros métodos. Aunque el riesgo de contaminación puede ser mayor debido a los dos ciclos de amplificación, esta técnica aumenta el rendimiento y la especificidad de la amplificación del ADN objetivo (Rahman et al., 2013).
El método de PCR anidada es una prueba de doble confirmación, ya que, si el primer cebador se une y amplifica una secuencia de ADN no deseada, el segundo conjunto de cebadores también se unirá dentro de la región no deseada.
Klemsdal y Elen (2006) detectaron Fusarium culmorum en matrices de piensos de cultivos de cereales donde el par de cebadores externos (FculAF, FculAR) se diluyó 10.000 veces en comparación con el par de cebadores internos (FculBF, FculBR) con un límite de detección de 5-50fg de ADN objetivo.
F.verticillioides productor de fumonisina asociado a cereales de matriz alimentaria se detectó utilizando un cebador directo (VERF-1) y dos inversos (VERTR y VERTF-2) (Deepa et al., 2016b).
PCR Anidada

Ventajas Desventajas
Método que requiere menos tiempo y que evita el aislamiento frecuente del ADN, para cada prueba con una amplificación precisa con menos concentración.
Mayor riesgo de contaminación.
12
METODOLOGÍA APLICACIÓN
PCR EN TIEMPO REAL (qPCR)
Este método detecta los amplicones de las moléculas de ADN objetivo basándose en la emisión de señales fluorescentes, lo que elimina los pasos de post-amplificación necesarios en el método de PCR convencional, lo que reduce el tiempo invertido. Detecta pequeñas concentraciones y es útil incluso para identificar en infecciones asintomáticas.
RT-PCR
METODOLOGÍA
Esta técnica se desarrolló para distinguir los hongos vivos de los muertos, ya que el ARNm se degrada rápidamente en las células muertas. Durante el proceso, el ARNm se transcribe de forma inversa para producir ADNc como plantilla mediante una enzima transcriptasa inversa (Brown et al., 2011).

Aunque puede ayudar a detectar infecciones asintomáticas, la tecnología qPCR no es eficaz cuando se aplica a los piensos (Ginzinger, 2002).
Ventajas Desventajas
Método eficaz en cuanto al tiempo, sensible y específico.
APLICACIÓN
Se detectó F. graminearum mediante el método RT-PCR en una matriz de piensos. El método es específico y altamente sensible para detectar hongos micotoxigénicos en los piensos. Sin embargo, también es más caro y laborioso que cualquier otro método (Guo et al., 2005).
Ventajas Desventajas
Equipos caros y necesidad de reactivos costosos y experiencia para manejar los instrumentos.
Es un método rápido y sencillo que no requiere gel de agarosa y permite obtener datos cualitativos y cuantitativos.
La técnica requiere de una mayor experiencia en el manejo de los instrumentos y es extremadamente sensible con un mayor riesgo de contaminación.
13
METODOLOGÍA
Métodos basados en la PCR
PCR-RFLP
Este método es útil para discriminar a nivel de especie con precisión, sensibilidad y especificidad.
El RFLP registra la variabilidad genética entre las distintas y mismas especies mostrando una diferenciación genética que se corresponde con los perfiles patógenos y micotoxicológicos.
Se detectaron especies de Aspergillus productoras de aflatoxinas, como A. flavus y A. parasiticus, en matrices complejas de alimentos y piensos (Ahmad et al., 2014).

Se describió la diferenciación por ITS-RFLP entre F. verticillioides y F. proliferatum (Visenti et al., 2012).
El método PCR-RFLP se aplicó para detectar variaciones entre 33 cepas de F. verticillioides productoras de fumonisina procedentes de matrices de piensos (Patino et al., 2006).
Se estudiaron las variaciones genéticas entre diez cepas de F. verticillioides utilizando seis enzimas diferentes (EcoRI, Xho I, Hae III, Kpn I, Hinf I, Hind III) aisladas de muestras de cereales (Deepa et al., 2018).
RFLP para Fusarium verticillioides
Ventajas Desventajas
Método sencillo, reproducible, rápido y estable que es preciso en la identificación de las poblaciones.
Laborioso y necesita de isótopos radioeléctricos.
14
APLICACIÓN
METODOLOGÍA
APLICACIÓN
PCR-AFLP
El polimorfismo de longitud de fragmento amplificado-PCR es una técnica diseñada para detectar polimorfismos de ADN.
METODOLOGÍA
La caracterización micotoxicológica y de patógenos fúngicos puede complementarse con el fingerprinting. El método es altamente reproducible, sensible y tiene la capacidad de amplificar 100 fragmentos a la vez.
PCR-RAPD

El ADN polimórfico amplificado al azar identifica las diferencias genotípicas utilizando cebadores de oligonucleótidos cortos para distinguir las especies de hongos micotoxigénicos y las variaciones dentro de la especie (Zhang et al., 2018).
Aunque el método se utiliza como herramienta de diagnóstico en los laboratorios, no se usa mucho en la práctica ya que carece de reproducibilidad (Blackwell et al., 1999).
El método se aplicó para estudiar la variabilidad genética entre 3.840 granos de maíz en Brasil y FUM1 y FUM8 fueron los genes esenciales responsables de la biosíntesis de fumonisina entre las especies de F. verticillioides (Silva et al., 2017).
Ventajas Desventajas
Alta reproducibilidad y posibilidad de analizar diferentes partes del genoma.
APLICACIÓN
Se identificaron F. subglutinans y F. verticillioides en granos de maíz basándose en las secuencias RAPD de los fragmentos (Moller et al., 1999).
El método RAPD-PCR se aplicó en diez cepas de F. verticillioides asociadas a muestras de cereales para estudiar la variación genética entre ellas (Deepa et al., 2018).
RAPD para especies de Fusarium
Protocolo complejo y largo.
Ventajas Desventajas
Método sencillo, eficaz y rápido que no requiere sondas.
Falta de reproducibilidad y posibles desajustes.
15
METODOLOGÍA
Métodos acoplados a PCR
PCR in situ
Esta técnica es una combinación de PCR e hibridación in situ para detectar hongos micotoxigénicos en tejidos o células (Long, 1998) que se ha convertido en algo común para analizar la expresión de genes e identificar ADN oARN en las células (Chu et al., 2019). Por lo general, la amplificación del ADN de las materias primas conduce a una interpretación errónea si el material contiene más de una especie.
Ventajas Desventajas
Se pueden aprovechar al máximo los tejidos pequeños, extensamente utilizados gracias a la microscopía optica.
Método que requiere mucho tiempo y es difícil de realizar con un número menor de copias de ADN/ ARN.
METODOLOGÍA
PCR-DGGE
Técnica que detecta comunidades fúngicas mediante la amplificación del ADN diana, inicialmente mediante PCR, y posteriormente sometida a electroforesis, pero que no sirve para describir las especies presentes en la muestra (Laforgue et al., 2009).
APLICACIÓN
Con este método se detectaron esporas de Trichoderma harzianum procedentes de matrices de alimentos. El método se aplica principalmente en estudios de diversidad y para detectar hongos patógenos, aunque es una técnica que requiere mucho tiempo y es poco reproducible (Rytkonen et al., 2011).

Ventajas Desventajas
Útil en los estudios de diversidad. Falta de reproducibilidad.
16
METODOLOGÍA
Métodos de postamplificación
LAMP
La amplificación isotérmica mediada por bucle (Loop-mediated isothermal amplification) es una técnica altamente sensible y exitosa que se aplica directamente in situ para garantizar la calidad y la seguridad de los alimentos, amplificando el ADN objetivo en menos de una hora a una temperatura constante mediante el uso de cuatro cebadores para identificar seis regiones en el ADN diana con especial atención en el paso de recocido (Tomita et al., 2008).
APLICACIÓN
Se detectó rápidamente Penicillium nordicum productor de ocratoxina directamente in situ en cadenas de producción de alimentos y piensos (Ferrara et al., 2015).
Ventajas Desventajas
Método rápido y muy específico. Alto riesgo de contaminación.
METODOLOGÍA
APLICACIÓN
ARRAYS DE ADN
La tecnología de arrays de ADN se desarrolló para detectar un gran número de organismos a la vez. Incluye las técnicas de microarrays y macroarrays, en las que se fijan secuencias detectoras específicas de cada especie, colocadas de forma diferenciada, en filtros de nailon o en un portaobjetos de cristal. Este método se aplica principalmente en los estudios de expresión génica.
Los microarrays consisten en un chip de ADN para analizar miles de ARNm simultáneamente, se utilizan principalmente para estudios de expresión génica y se aplican para la identificación de especies de Aspergillus (Singh y Kumar, 2013).
Los macroarrays implican una técnica de hibridación de ADN y se utilizan principalmente para la identificación de A.alternata, F. solani y A. fumigatus en matrices de alimentos (Singh y Kumar, 2013).
Kristensen et al. (2007) aplicaron la tecnología de microarrays para la detección de especies de Fusarium, lo que parece ser fácil, seguro, rápido y rentable en matrices de piensos y alimentos. La detección de especies de Fusarium que producen 14 tricotecenos y moniliformina se determinó mediante un microarray de ADN.
Ventajas Desventajas
Análisis rápido, fácil y a granel que no requiere una gran cantidad de muestra de ADN.
Técnica cara que requiere más tiempo y más cantidad de ARNm.
17
METODOLOGÍA
Nuevas tecnologías emergentes para la detección de micotoxinas
Además de los métodos cromatográficos (cromatografía en capa fina, cromatografía de gases y cromatografía líquida) y de los métodos basados en inmunoensayos (membranas de flujo, ELISA, tiras reactivas, inmunoensayo de polarización de fluorescencia (FPI) e inmunoensayo de flujo lateral (LFA)) (Agriopoulou et al., 2020), se pueden aplicar muchas otras técnicas emergentes para la detección de micotoxinas en los piensos.
BIOSENSORES
Este método de ensayo in situ, rápido, fácil, reproducible, estable, preciso y barato, requiere transductores (ópticos, piezoeléctricos oelectroquímicos) para la detección de micotoxinas (Schulz et al., 2019).
APLICACIÓN
Oliveria et al. (2019) reportaron el uso de biosensores para la detección de micotoxinas producidas por hongos micotoxigénicos en piensos y alimentos.

El método detectó AFB1, OTA, AFM1, PAT, ZEN, FB1, T-2 y DON en polvo de maíz, maíz, cebada, trigo, maíz y productos de maíz (Hossain et al., 2019).
Los sensores impedimétricos detectaron hongos micotoxigénicos productores de AFB1, AFM1, OTA y PAT en alimentos y piensos (Khan et al., 2019).
Se utilizaron sensores fotométricos con un sistema de dos o tres electrodos para analizar con éxito la AFB1 y el ZEN en el polvo de maíz y el maíz, respectivamente (He et al., 2019).
También se han utilizado sensores amperométricos basados en un sistema de dos o tres electrodos para detectar el ZEN en el maíz y los productos a base de maíz (He et al., 2019).
Los sensores de resonancia de plasmón superficial son sensibles y rentables con una alta especificidad para detectar DON, ZEN y la toxina T-2 en el trigo (Hossain et al., 2019).
Los sensores de microbalanza de cristal de cuarzo detectaron AFB1 en arroz y trigo (Gu et al., 2019).
Ventajas Desventajas
Técnica fácil de usar, in situ, con buena sensibilidad y selectividad. Alta rentabilidad.
18
METODOLOGÍA
APLICACIÓN
NARIZ ELECTRONICA
La nariz electrónica incluye detectores no específicos que capturan compuestos orgánicos volátiles (VOCs) y detectan huellas volátiles cualitativas asociadas a los hongos toxigénicos.
METODOLOGÍA
El olor es la información preliminar para detectar los metabolitos producidos en la identificación de VOCs específicos relacionados con los hongos en las matrices de los piensos.
POLARIZACIÓN FLUORESCENTE (PF)
La PF trabaja con el analito y el trazador en el sitio de unión del anticuerpo. La unión del trazador al anticuerpo inluye en la rotación de la molécula del trazador aumentando el valor de la PF.
La cantidad de trazador es inversamente proporcional al analito libre en la muestra, lo que resulta en un valor de polarización inversamente proporcional a la concentración de analito.
El método detecta las micotoxinas producidas por hongos micotoxigénicos, como las aflatoxinas, las fumonisinas en el maíz y el DON en el salvado de trigo, que es el principal ingrediente de los piensos (Ottoboni et al., 2018).
Ventajas Desventajas
Se puede detectar un bajo nivel de micotoxinas si se optimiza.
APLICACIÓN
El método detecta las especies de Fusarium que producen ZEN en el maíz y DON en los productos a base de trigo, así como las especies de Aspergillus que producen AFB1 en el maíz, y OTA en el arroz (Huang et al., 2020).
El método es preciso en comparación con el HPLC con una sensibilidad limitada (Zhang et al., 2018).
La naturaleza no volátil de las micotoxinas es problemática para la detección a través de una nariz electrónica.
Ventajas Desventajas
Técnica rápida y precisa sin pasos preanalíticos.
Sensibilidad limitada y se requiere experiencia para manejar el instrumental.
19
RMN CUANTITATIVA E IMÁGENES HIPERESPECTRALES
La espectroscopia de resonancia magnética nuclear (RMN) se utiliza para la identificación de compuestos orgánicos y la metabolómica. Recientemente, se ha aplicado la RMN 2D y multidimensional.
METODOLOGÍA APLICACIÓN
La imagen hiperespectral es una técnica para la evaluación de micotoxinas fúngicas que funciona con menos costes sin destruir la muestra, siendo rápida y operando en la localización de datos espectrales.
La metabolómica basada en la RMN se ha utilizado para dilucidar el mecanismo de reordenación de la micotoxina de Fusarium, Fusarina C, en los piensos debido a su alto rendimiento de las muestras y a la metabolómica con rica información estructural (López-Ruiz et al., 2019).

Las imágenes hiperespectrales se han utilizado principalmente para detectar la contaminación por DON y patógenos de Fusarium en los granos de trigo a granel (Femenias et al., 2020).
Ventajas Desventajas
Los compuestos se pueden dilucidar sin estándares y se requiere una mínima preparación de la muestra.
Límite de detección más bajo, baja sensibilidad y mayores requisitos de experiencia.
20
CONCLUSIONES
Algunas materias primas agrícolas, como el trigo, el maíz, la soja, el mijo perla, el sorgo y el arroz, se utilizan principalmente para la fabricación de piensos, que son los más demandados por las industrias ganaderas.
Por ello, la seguridad de los piensos es fundamental, ya que su consumo es una vía potencial de entrada de sustancias químicas peligrosas en la cadena alimentaria humana, incluidas las micotoxinas.
Debido a la amenaza que suponen las micotoxinas para la salud animal y humana, en la UE se han aplicado determinadas legislaciones a los productos basados en piensos destinados al ganado con el establecimiento de criterios de limitación verificados, así como el desarrollo de métodos sensibles y coherentes para detectar las micotoxinas.
Para detectar las micotoxinas y los hongos micotoxigénicos se aplican tecnologías basadas en la PCR y técnicas analíticas novedosas.
Son rápidas, rentables, precisas, sensibles, portátiles y seguras, para la detección en matrices de alimentos y piensos.
En la actualidad, existen muchos más métodos moleculares, como NASBA, FISH, SAGE, Northern blotting, método basado en sondas de ADN/ARN, PCR-ELISA, MCH-PCR, PCR de próxima generación, PCR cooperativa, ARN de interferencia. Sin embargo, aún no se han aplicado para la detección de hongos micotoxigénicos asociados a matrices de alimentos y piensos.
Diferentes países han establecido ciertas normas y regulaciones para los niveles de toxinas en matrices de alimentos y piensos. Los recientes avances en tecnologías novedosas, como la nariz electrónica, la polarización fluorescente, los MIPs, los biosensores y los métodos moleculares, revelan las posibilidades en la determinación de micotoxinas y se proponen como las futuras técnicas de análisis independientes para las empresas de piensos y alimentos.
Por lo tanto, es importante desarrollar técnicas para la detección in situ de toxinas manteniendo la especificidad y la sensibilidad.
21
REFERENCIAS
1. Agriopoulou, S., Stamatelopoulou, E., Varzakas, T. 2020. Advances in analytical and detection of major mycotoxins in foods. Foods, 9, 518.
2. Ahmad M.M., Ahmad M., Ali A., Hamid R., Javed S., Abdin M.Z., 2014. Detection of Aspergillus flavus and Aspergillus parasiticus from aflatoxin-contaminated peanuts and their differentiation using PCR-RFLP. Annals of Microbiology 64: 1597-1605.
3. Bindslev L., Oliver R.P., Johansen, B., 2002. In situ PCR for detection and identification of fungal species. Mycological Research 106: 277-279.
4. Blackwell B.A., Gilliam J.T., Savardm M.E., Miller D., Duvick J.P. 1999. Oxidative deamination of hydrolysed fumonisin B1 (AP1) by cultures of Exophiala spinifera. Natural Toxins. 7: 31–38.
5. Brown N.A., Bass C., Baldwin T.K., Chen H., Massot F., Carion P.W., Urban M., Van De Meene A.M., Hammond-Kosack K.E. 2011. Characteri sation of the Fusarium graminearum in wheat and floral inter- action. Journal of Pathogens.
6. CAST. 2003. Mycotoxins: Risks in Plant, Animal, and Human Systems; CAST: Ames, IA, USA.
7. Chu Y.H., Hardin, H., Zhang, R., Guo, Z. and Lloyd, R.V., in press. In situ hybridization: introduction to techniques, applications and pitfalls in the performance and interpretation of assays. Seminars in Diagnostic Pathology. 36: 336-341.
8. Dass R.S., Sreenivasa M.Y., Janardhana G.R. 2007. High incidence of Fusarium verticillioides in Animal and Poultry feed mixtures produced in Karnataka, India. Plant Pathology Journal. 6:174-178.
9. Dass R.S., Sreenivasa M.Y., Charith Raj A.P., Janardhana G.R. 2009. PCR-based assay for the rapid detection of fumonisin-producing Fusarium species in maize-based animal and poultry feeds in Karnataka, India. Arhieves of Phytopathology and plant protection. 42:8, 796-804
10. Deepa N., Charith Raj Adkar-Purushothama., Sreenivasa M.Y. 2016b. Nested PCR method for early detection of fumonisin producing Fusarium verticillioides in pure cultures, cereal samples and plant parts. Food Biotechnology 30: 18-29.
11. Deepa N., Charith Raj Adkar-Purushothama., Sreenivasa M.Y. 2016c. Multiplex PCR for the early detection of fumonisin producing Fusarium verticillioides. Food Bioscience. 13: 84-88.
12. Deepa N., Nagaraja H., Sreenivasa, M.Y. 2016a. Prevalence of fumonisin producing Fusarium verticillioides associated with cereals grown in Karnataka (India). Food Science and Human Wellness, 5(3), 156-158.
13. Deepa N., Rakesh S and Sreenivasa M.Y. 2018. “Morphological, pathological and mycotoxicological variations among Fusarium verticillioi des isolated from cereals”. 3 biotech. 8:105.
14. Deepa N., Sreenivasa M.Y. 2017b. “Fusarium verticillioides, a globally important pathogen of Agriculture and Livestock: A review”. Journal of Veterinary Medicine and Research. 4(4): 1084.
15. Deepa, N., Charith Raj Adkar-Purushothama, Sreenivasa, M.Y. 2021. Chapter 6: “Molecular technologies for the early detection of fungal phytopathogens associated with cereal crops”. Microbial genomic technologies to combat the problems of farming lands. Microbes in land use change management. Elsevier. 137-154.
16. Deepa, N., Sreenivasa, M.Y. 2017a. “Fumonisins: A review on its Global Occurrence, Epidemiology, Toxicity and Detection”. Journal of Veterinary Medicine and Research. 4(6): 1093.
17. Deepthi, B.V., Gnanaprakash, A.P., Sreenivasa M.Y. 2017. Effet of γ-irradiation on fumonisin producing Fusarium associated with animal and poultry feed mixtures. 3 Biotech. 7:57.
18. Deepthi, B.V., Sreenivasa M.Y. 2020. Fumonisins - The underrated mycotoxins in poultry, livestock and humans. mycotoxinsite. Com
19. Dinolfo, M., Barros, G. and Stenglein, S., 2012. Development of a PCR assay to detect the potential production of nivalenol in Fusarium poae. FEMS Microbiology Letters 332: 99-104.
20. EU Commission. 2015. The European Parliament and The Council of the European Union Directive 2002/32/EC of 22 the European Parliament and of the Council of 7 May 2002 on undesirable substances in animal feed. Official Journal of the European Union. 32, 1–30.
21. European Commission. Comission recomendation of 14 January 2011 establishing guidelines for the distinction between feed materials, feed additives, biocidal products and veterinary medicinal products. 2011. Official Journal of the European Union. 75–79.
22. FAO & WHO. 2007. Animal Feed Impact on Food Safety; FAO: Rome, Itlay,
22
23. FAO. 2009. The State of Food and Agriculture; FAO: Rome, Italy. ISBN 9789251062159.
24. Faria C.B., Abe C.A.L., Novais da- silva C., Tessmann D.J., Barbosa-Tesmann L.P. 2012. New PCR assays for the identification of Fusarium verticillioides, F. subglutinans, and other species of the Gibberella fujikuroi complex. International Journal of Molecular Sciences. 13: 115-132.
25. Femenias, A., Gatius, F., Ramos, A.J., Sanchis, V., Marín, S. 2020. Use of hyperspectral imaging as a tool for Fusarium and deoxynivalenol risk management in cereals: A review. Food Control. 108, 106819.
26. Ferrara M., Perrone G., Gallo A., Epifani F., Visconti A., Susca A., 2015. Development of loop-mediated isothermal amplification (LAMP) assay for the rapid detection of Penicillium nordicum in dry- cured meat products. International Journal of Food Microbiology. 202: 42-47.
27. Food Standards Agency Food.gov.uk. Available online: https://www.food.gov.uk/business-industry/farmingfood/animalfeed/what-farmanimals-eat (accessed on 4 December 2016).
28. Ginzinger D.G. 2002. Gene quantification using real-time quantitative PCR: An emerging technology hits the mainstream. Experimental Hematology. 30: 503-512.
29. Gu, Y., Wang, Y., Wu, X., Pan, M., Hu, N., Wang, J., Wang, S. 2019. Quartz crystal microbalance sensor based on covalent organic framework composite and molecularly imprinted polymer of poly(o-aminothiophenol) with gold nanoparticles for the determination of aflatoxin B1. Sensors and Actuators B Chemistry. 291, 293–297.
30. Guo J.R., Schnieder F., Beyer M., Verreet, J.A., 2005. Rapid detection of Mycosphaerella graminicola in wheat using reverse transcrip tion-PCR assay. Journal of Phytopathology 153: 674-679.
31. He, B., Yan, X. 2019. An amperometric zearalenone aptasensor based on signal amplification by using a composite prepared from porous platinum nanotubes, gold nanoparticles and thionine-labelled graphene oxide. Microchimica Acta 2019, 186, 383.
32. Hossain, Z., Busman, M., Maragos, C.M. 2019. Immunoassay utilizing imaging surface plasmon resonance for the detection of cyclopiazonic acid (CPA) in maize and cheese. Analytical and Bioanalytical Chemistry. 411, 3543–3552.
33. Huang, X., Tang, X., Jallow, A., Qi, X., Zhang, W., Jiang, J., Li, H., Zhang, Q., Li, P. 2020. Development of an Ultrasensitive and Rapid Fluorescence Polarization Immunoassay for Ochratoxin A in Rice. Toxins. 12, 682.
34. Khan, R., Ben Aissa, S., Sherazi, T.A., Catanante, G., Hayat, A., Marty, J.L. 2019. Development of an Impedimetric Aptasensor for Label Free Detection of Patulin in Apple Juice. Molecules. 24, 1017.
35. Kim S.W., Li Z., Moore P.S., Monaghan A.P., Chang Y., Nichols M., John B. 2010. A sensitive non-radioactive northern blot method to detect small RNAs. Nucleic Acids Research. 38: 98-110.
36. Klemsdal S.S., Elen O. 2006. Development of a highly sensitive nested PCR method using a single closed tube for detection of Fusarium culmorum in cereal samples. Letters in Applied microbiology. 42: 544-548.
37. Kristensen R., Gauthier G., Berdal K.G., Hamels S., Remacle J., Holst-Jensen A., 2007. DNA microarray to detect and identify trichotheceneand moniliformin-producing Fusarium species. Journal of Applied Microbiology 102: 1060-1070.
38. Laforgue R., Guérin L., Pernelle J.J., Monnet C., Dupont J., Bouix M., 2009. Evaluation of PCR-DGGE methodology to monitor fungal communities on grapes. Journal of Applied Microbiology 107: 1208-1218.
39. Long A.A. 1998. In situ polymerase chain reaction: foundation of the technology and today’s options. European Journal of Histochemistry 42: 101-109.
40. López-Ruiz, R., Romero-González, R., Frenich, A.G. 2019. Metabolomics approaches for the determination of multiple contaminants in food. Current Opinion in Food Science. 28, 49–57.
41. Moeller E.M., Chelkowski J., Geiger H.H. 1999. Species-specific PCR assays for the fungal pathogens Fusarium moniliforme and Fusarium subglutinans and their application to diagnose maize ear rot. Journal of Phytopathology. 147: 497-508.
42. Oliveira, I.S., Junior, A.G.D.S., De Andrade, C.A.S., Oliveira, M.D.L. 2019. Biosensors for early detection of fungi spoilage and toxigenic and mycotoxins in food. Current Opinion in Food Science. 29, 64–79.
43. Ottoboni, M., Pinotti, L., Tretola, M., Giromini, C., Fusi, E., Rebucci, R., Grillo, M., Tassoni, L., Foresta, S., Gastaldello, S. et al. 2018. Combining E-Nose and Lateral Flow Immunoassays (LFIAs) for Rapid Occurrence/Co-Occurrence Aflatoxin and Fumonisin Detection in Maize. Toxins. 10, 416.
23
44. Pereira, C.S., Cunha S.C., Fernandes, J.O. 2019. Prevalent mycotoxins in animal feed: occurrence and analytical methods. Toxins. 11, 290.
45. Porter-Jordan, K., Rosenberg, E.I., Keiser, J.F., Gross, J.D., Ross, A.M., Nasim, S. and Garrett, C.T., 1990. Nested polymerase chain reaction assay for the detection of cytomegalovirus overcomes false positives caused by contamination with fragmented DNA. Journal of Medical Virology 30: 85-91.
46. Priyanka S.R., Mudili D.V., Balakrishna K.S., Murali H., Batra H., 2015. Development and evaluation of a multiplex PCR assay for simultaneous detection of major mycotoxigenic fungi from cereals. Journal of Food Science and Technology 52: 486-492.
47. Rahman H, Ur., Yue X., Yu Q., Zhang W., Zhang Q., Li P. 2020. Current PCR-based methods for the detection of mycotoxigenic fungi in complex food and feed matrices. World Mycotoxin Journal, 13 (2): 139-150.
48. Ramana M.V., Balakrishna K., Murali H.C.S., Batra H.V. 2011. Multiplex PCR based strategy to detect contamination with mycotoxigenic Fusarium species in rice and finger millet collected from southern India. Journal of the Science of Food and Agriculture. 91: 1666-1673.
49. Rashmi R., Ramana M.V., Shylaja R., Uppalapati S.R., Murali H.S., Batra H.V., 2013. Evaluation of a multiplex PCR assay for concurrent detection of four major mycotoxigenic fungi from foods. Journal of Applied Microbiology 114: 819-827.
50. Rytkönen K.T., Williams T.A., Renshaw G.M., Primmer C.R., Nikinmaa M. 2011. Molecular evolution of the metazoan PHD-HIF oxygen-sensing system. Molecular Biology and Evolution. 28: 1913–1926
51. Schulz, K., Pöhlmann, C., Dietrich, R., Märtlbauer, E., Elßner, T. 2019. Electrochemical Biochip Assays Based on Anti-idiotypic Antibodies for Rapid and Automated On-Site Detection of Low Molecular Weight Toxins. Frontiers in Chemistry. 7, 31.
52. Silva J.J., Viaro H.P., Ferranti L.S., Oliveira A.L.M., Ferreira J.M., Ruas C.F., Ono E.Y.S., Fungaro M.H.P. 2017. Genetic structure of Fusarium verticillioides populations and occurrence of fumonisins in maize grown in southern Brazil. Crop Protection. 99: 160-167.
53. Singh A., Kumar N., 2013. A review on DNA microarray technology. International Journal of Current Research and Reviews. 5: 01–05.
54. Smith M.C., Madec S., Coton E., Hymery N. 2016. Natural Co-occurrence of mycotoxins in foods and feeds and their in vitro combined toxicological effects. Toxins. 8, 94.
55. Sreenivasa M.Y., Dass R.S., Charithraj A.P., Janardhana G.R., 2007. PCR method for the detection of genus Fusarium and fuonisin producing isolates from freshly harvested sorghum grains grown in Karnataka, India. J food saf. 28, 236-247.
56. Sreenivasa M.Y., Gonzalez Jaen M.T., Dass R.S., Charithraj A.P., Janrdhana G.R., 2008. A PCR based assay for the detetction and differentia tion of potential fumonisin producing Fusarium verticillioides isolated from maize kernels. Food biotechnol. 22, 160-170.
57. Tomita N., Mori Y., Kanda H., Notomi T., 2008. Loop-mediated isothermal amplification (LAMP) of gene sequences and simple visual detection of products. Nature Protocols 3: 877-882.
58. Visentin I., Montis V., Doll K., Alabouvette C., Tamietti G., Karlovsky P., Cardinale F. 2012. Transcription of genes in biosynthetic pathway for fumonisin mycotoxins is epigenetically and differentially regulated in the fungal maize pathogen Fusarium verticillioides. Eukaryotic cell. 11: 252-259.
59. Zhang, X., Tang, Q., Mi, T., Zhao, S., Wen, K., Guo, L., Mi, J., Zhang, S., Shi, W., Shen, J., et al. 2018. Dual-wavelength fluorescenc polarization immunoassay to increase information content per screen: Applications for simultaneous detection of total aflatoxin and family zearalenones in maize. Food Control. 87, 100–108.
60. Zhao Y., Liu N., Niu, J.X. 2009. A study of the distribution of apple stem pitting virus in tissues of pear tree using in situ hybridization and in situ RT-PCR. Agricultural Sciences in China 8: 1351-1359.
24