


Marilyn J. Roossinck




Marilyn J. Roossinck



Die faszinierende Welt unserer heimlichen Mitbewohner







Marilyn J. Roossinck
Die faszinierende Welt unserer heimlichen Mitbewohner
Übersetzt von Coralie Wink und Monika Niehaus

Haupt Verlag

1.Auflage: 2025
ISBN 978-3-258-08407-7
Alle Rechte vorbehalten.
Copyright © 2025 für die deutschsprachige Ausgabe: Haupt Verlag, Bern
Jede Art der Vervielfältigung ohne Genehmigung des Verlages ist unzulässig.
Aus dem Englischen übersetzt von Coralie Wink, D-Dossenheim, und Monika Niehaus, D-Düsseldorf
Satz der deutschsprachigen Ausgabe: Die Werkstatt Medien-Produktion GmbH, D-Göttingen
Umschlagabbildungen: Kateryna Kon/Shutterstock
Die englischsprachige Originalausgabe erschien 2023 unter dem Titel Viruses – A Natural History bei Princeton University Press, USA.
Copyright © 2023 UniPress Books Limited
Gedruckt in Malaysia
Um lange Transportwege zu vermeiden, hätten wir dieses Buch gerne in Europa gedruckt. Bei Lizenzausgaben wie diesem Buch entscheidet jedoch der Originalverlag über den Druckort. Der Haupt Verlag kompensiert mit einem freiwilligen Beitrag zum Klimaschutz die durch den Transport verursachten CO2-Emissionen. Dabei unterstützt der Verlag ein Projekt zur nachhaltigen Forstbewirtschaftung in der Zentralschweiz. Wir verwenden FSC®-zertifiziertes Papier. FSC® sichert die Nutzung der Wälder gemäß sozialen, ökonomischen und ökologischen Kriterien.
Diese Publikation ist in der Deutschen Nationalbibliografie verzeichnet. Mehr Informationen dazu finden Sie unter http://dnb.dnb.de.
Der Haupt Verlag wird vom Bundesamt für Kultur für die Jahre 2021–2025 unterstützt.
Sie möchten nichts mehr verpassen?
Folgen Sie uns auf unseren SocialMedia-Kanälen und bleiben Sie via Newsletter auf dem neuesten Stand.
www.haupt.ch/informiert
Wir verlegen mit Freude und großem Engagement unsere Bücher. Daher freuen wir uns immer über Anregungen zum Programm und schätzen Hinweise auf Fehler im Buch, sollten uns welche unterlaufen sein.
Haupt Verlag AG, Falkenplatz 14, CH-3012 Bern verlag@haupt.ch
www.haupt.ch




































160
26
DIE VIELFALT VON VIREN
62
WIE VIREN SICH VERMEHREN
104
WIE VIREN SICH VERBREITEN
134
EVOLUTION

DER KAMPF ZWISCHEN VIREN UND IHREN WIRTEN
194
DIE FUNKTION VON VIREN IN ÖKOSYSTEMEN
220
DIE GUTEN VIREN
248
DIE PATHOGENE
278 Glossar

280 Quellen
282 Register
288 Bildnachweis






Viren sind seit dem Auftreten eines neuartigen Coronavirus Ende 2019/Anfang 2020 schlagartig ins Licht der Öffentlichkeit gerückt. Es handelte sich um das severe acute respiratory syndrome coronavirus 2 (Schweres-aktues-respiratorisches-SyndromCoronavirus-2, SARS-CoV-2). Plötzlich wurde uns bewusst, wie sehr Viren unser Leben beeinflussen können! Die Medien und selbst die virologische Fachwelt wurden von den detaillierten Informationen über dieses Virus – das die Coronavirus-Krankheit-2019 (coronavirus disease 2019, COVID-19) verursacht – überwältigt (oder überfordert). SARS-CoV-2 hatte und hat weltweit immense Auswirkungen, doch dies ist nur ein winziger Ausschnitt aus der Welt der Viren. Dieses Buch lädt Sie zu einer faszinierenden Reise ein, die weit über COVID-19 hinausgeht und ins Reich der vielfältigsten biologischen Einheiten unserer Erde führt.
Aufgrund der großen Vielfalt der Viren ist es gar nicht so einfach, eine Definition zu finden, die sämtlichen Virustypen gerecht wird.
Es gibt allerdings einige Merkmale, die allen Viren gemeinsam sind: Ihr Genom besteht aus RNA oder DNA, sie benötigen für all ihre Funktionen einen Wirt und sie besitzen das genetische Material für zahlreiche komplexe Funktionen, sind aber nicht in der Lage, ihre eigene Energie zu erzeugen.
Ob Viren lebendig sind oder nicht, wird nach wie vor kontrovers diskutiert: Als Viren entdeckt wurden, hielt man sie für lebendig; als man aber 1935 das Tabakmosaikvirus als Kristall darstellen konnte, waren Teile der Wissenschaft der Meinung, sie seien eher eine chemische Substanz als eine Lebensform. Manche Virolog:innen vertreten die Ansicht, dass Viren lebendig sind, wenn sie eine Wirtszelle infizieren, außerhalb einer Zelle aber eher mit Samen oder Sporen verglichen werden können..
Kurzum, es gibt keine einfache Antwort auf die Frage, ob Viren lebendig sind. Für und wider gab und gibt es viele Argumente, die aber selten von Virolog:innen vorgebracht werden. Denn Virolog:innen finden die Objekte ihres Interesses generell faszinierend – da macht es wenig aus, ob lebendig oder nicht, denn Viren beeinflussen auf jeden Fall das Leben aller Organismen auf dieser Erde.
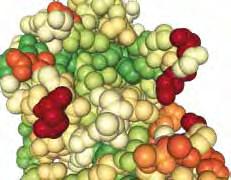
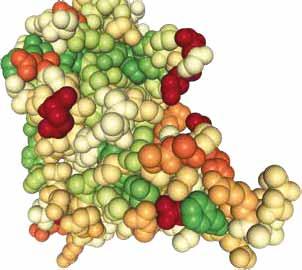
Hochauflösende Kryo-EMStruktur des Zikavirus.

Eine Zelle ist die Grundeinheit des Lebens. Es gibt zwei Gruppen von Zellen: prokaryotische und eukaryotische. Zu den Prokaryoten zählen Bakterien und Archaea, die meist aus Einzelzellen bestehen – einige können jedoch vielzellige Strukturen bilden. Alle anderen Organismen sind Eukaryoten.
Die Grundlagen des zellulären Lebens
Alles Leben besteht aus Zellen, die entweder prokaryotisch oder eukaryotisch sind. Die Zellen von Bakterien und Archaeen sind prokaryotisch. Sie haben keinen Zellkern und sind normalerweise von einer Zellwand umgeben. Tier- und Pflanzenzellen sind eukaryotisch: Sie haben einen Kern, der das Genom des Organismus beherbergt. Tierische Zellen besitzen keine Zellwand, die meisten anderen eukaryotischen Zellen hingegen schon. Die Strukturen innerhalb von eukaryotischen Zellen werden als Organellen bezeichnet und sind von ihrer eigenen Membran umgeben. Die Energie der Zellen wird in den meisten eukaryotischen Zellen von Mitochondrien, in den Pflanzenzellen ferner von Chloroplasten gewonnen; beide leiten sich ursprünglich von Bakterien ab. Mitochondrien und Chloroplasten haben ihr eigenes (DNA-)Genom, können außerhalb der Eukaryotenzelle aber nicht überleben. Zellen werden in der Durchschnittsgröße für den jeweiligen Zelltyp dargestellt, tatsächlich variiert die Größe enorm. Volumenmäßig ist die Eizelle eines Straußeneis die größte bekannte Zelle.
Künstlerische Darstellung eines kurzen Stücks einer DNA-Doppelhelix.

BAKTERIENZELLE (PROKARYOTISCH)
Ribosom
Nucleoid (DNA)
Cytoplasma
Zellmembran
Zellwand
Kapsel
Pilus
Flagellum
raues endoplasmatisches Retikulum
Nucleus (Kern)
Nucleolus (Kernkörperchen)
Lysosom
Golgi-Apparat
Mitochondrium
Zellmembran
Centriol
glattes endoplasmatisches Retikulum
Kernmembran
Cytoplasma
Ribosom
Vakuole
Vakuole
Zellwand
Kernmembran
Nucleolus (Kernkörperchen)
Nucleus (Kern)
Cytoplasma
PFLANZENZELLE (EUKARYOTISCH)
Mitochondrium
Centriol
Chloroplast
raues endoplasmatisches Retikulum
Zellmembran
Ribosom
glattes endoplasmatisches Retikulum
Golgi-Apparat




Modell eines Querschnitts der Schwanzstruktur eines Sulfolobus-Spindle-Shaped-Virus 19, abgeleitet aus Kryo-EM-Daten.
Bicaudaviridae
Fuselloviridae
Spiraviridae
Tristromaviridae
Thaspiviridae
Bändermodell eines Sulfolobus-islandicus-Rod-Shaped-Virus, abgeleitet aus Kryo-EM-Daten. Der Wirt dieses Virus lebt in sehr saurem und heißem (80 °C) Wasser. Die DNA lässt sich in der Struktur als Helix erkennen, doch es handelt sich um eine andere DNAForm, die als A-Struktur bezeichnet wird. In extremen Habitaten ist diese Form der DNA stabiler als die gewöhnliche B-Struktur.
Strukturmodell des SulfolobusTurreted-Icoahedral-Virus, abgeleitet aus Kryo-EM-Daten.
Die enorme Variationsbreite von Formen bei Viren, die Archaeen infizieren.
Das Genom von Viren besteht entweder aus DNA oder RNA, die entweder doppelsträngig (ds) oder einzelsträngig (ss) vorliegen (siehe Tabelle S. 38/39). Virale Genome können linear oder ringförmig sein und ein Segment oder mehrere Segmente umfassen –ähnlich wie unser eigenes Genom, das in mehrere Segmente unterteilt ist, die wir Chromosomen nennen (wir haben einen doppelten Chromosomensatz mit je 23 Chromosomen).
Die Genomtypen der Viren beschränken sich auf die Infektion bestimmter Organismenreiche, wobei ssDNA (single stranded desoxyribonucleic acid) Typ II der einzige Typ ist, der in allen Reichen nachgewiesen ist. Bei den Archaeen findet man keine RNA-Viren und in Pflanzen außer Algen keine dsDNA-Viren Typ I. Die Gründe dafür sind nicht immer bekannt, doch einige stehen möglicherweise mit der Biologie des Wirts in Zusammenhang. Beispielsweise sind die meisten dsDNA-Viren (double stranded desoxyribonucleic acid) groß, und benachbarte Pflanzenzellen weisen nur sehr kleine Öffnungen zwischen einander auf, die zu eng sind, als dass große Viren hindurchschlüpfen könnten. Allgemein gesagt, haben Pflanzen die größten Zellen unter allen Lebewesen und weisen den größten Teil der kleinsten Viren auf.
Wie viele Viren gibt es überhaupt? Die ersten großen Untersuchungen der viralen Biodiversität Anfang der 1990er-Jahre beschäftigten sich mit Viren im Meer. Dabei wurden einfach Viruspartikel im Meerwasser gezählt, die mittels Elektronenmikroskopie oder Fluoreszenzmethoden sichtbar gemacht wurden. Schätzungen anhand dieser Daten kamen zu dem Ergebnis, dass es rund 1030 (eine 1 mit 30 Nullen) Viruspartikel im Meer gibt – oder 10 Millionen Mal so viele wie die Anzahl der Sterne im ganzen Universum. Auch wenn ein einzelnes Virus sehr klein ist, besitzen Viren insgesamt dennoch eine riesige Biomasse – sie entspricht
etwa dem 15-Fachen der Biomasse aller Wale in unseren Meeren in den Zeiten vor Beginn des Walfangs. Wenn wir als durchschnittliche Größe eines marinen Virus 100 nm annehmen und man alle zu einer Kette zusammenfügen könnte, würde diese Kette bis weit über die Milchstraße hinausreichen. Über landbewohnende (terrestrische) Viren wissen wir weit weniger. Obgleich viele terrestrische Systeme ebenfalls auf Viren untersucht wurden, ist es technisch schwierig, sie zu studieren. Die oben angegebenen Zahlen für marine Viren beziehen sich auf die Anzahl von geschätzten individuellen Viruspartikeln, doch wie viele unterschiedliche Virenarten gibt es dort? Die Antwort ist: Wir wissen es nicht. Ständig werden neue Viren identifiziert, doch das International Committee on Taxonomy of Viruses (ICTV, Internationales Komitee für die Taxonomie von Viren) erkennt bislang nur wenig mehr als 9000 Arten (Spezies) an. Das ist höchstwahrscheinlich nur ein kleiner Bruchteil der Anzahl, die tatsächlich auf unserem Planeten existiert.
Abbildungen von Viren aus verschiedenen Quellen, einschließlich künstlerischer Interpretationen und elektronenmikroskopischer Darstellungen, die eine Reihe von Formen zeigen:
A: Influenzavirus (Grippevirus); B: Cytomegalovirus; C: Variolavirus (Pockenvirus); D: Tollwutvirus.





Einige Viren können ihr Genom zwischen RNA und DNA umwandeln. Ein Beispiel sind die Retroviren, die ein einzelsträngiges (+)-RNA-Genom aufweisen und zwei Kopien davon im Viruspartikel tragen. Zudem besitzen sie eine einzigartige Transkriptase, die als Reverse Transkriptase bezeichnet wird. Dieses Enzym kann RNA in DNA umkopieren – ein Vorgang, den frühe Molekularbiolog:innen für unmöglich hielten, bis er in den 1970er-Jahren entdeckt wurde.
Retroviren gelangen in die Zelle, indem ihre Hüllmembran mit der zellulären Plasmamembran fusioniert. Im Zellinneren kopiert die Reverse Transkriptase das RNA-Genom zu doppelsträngiger DNA um. Diese DNA wird dann in den Zellkern und dort in das Genom der Wirtszelle integriert, wo sie den Rest des Lebens dieser Zelle und auch in deren Nachkommen verbleibt. Wenn das in einer Keimbahnlinie (d. h. in Samen- oder Eizellen) geschieht, wird das Virus zu einem permanenten Teil des Wirtsgenoms. Darum findet man so viele retrovirale Sequenzen in Genomen (acht Prozent des menschlichen Genoms sind retroviral). In der Regel infizieren diese Viren aber keine Keimbahnzellen, daher wird das integrierte Virus nicht an die Nachkommen des Wirts weitergegeben.
Sobald die retrovirale DNA in das WirtszellGenom integriert ist, agiert sie ganz ähnlich wie ein beliebiges anderes Gen. Die Wirtsenzyme stellen durch Ablesen des integrierten Virus Messenger-RNA her, die sie dann spleißen. Spleißen (splicing) ist ein sehr häufiger Vorgang bei der Prozessierung der Messenger-RNA. Zelluläre Messenger-RNA wird mit Teilen hergestellt: Introns werden entfernt, während Exons beibehalten werden. Die Introns werden von zellulären Enzymen herausgeschnitten, um die reife Messenger-RNA herzustellen. Selektives Spleißen sorgt dafür, dass die einzelne RNA in mehrere unterschiedliche
Messenger-RNAs umgewandelt werden kann, sodass sie sämtliche für das Virus notwendigen Proteine herstellen kann. Einige dieser Proteine sind Polyproteine, die von Proteasen in die erforderlichen funktionellen Einzelproteine zerschnitten werden. Diese speziellen Proteasen gibt es nur bei Retroviren, und sie sind Zielstrukturen für die Entwicklung antiretroviraler Medikamente. Auch das Virusgenom wird von der integrierten DNA hergestellt. Dieses Genom wird dann in neue Virenpartikel verpackt – jeweils zwei Genome pro Partikel. Die Core-Partikel des Virus knospen dann durch die Plasmamembran des Wirts und erhalten auf dem Weg aus der Zelle ihre neue Membranhülle. Retroviren dienen in der Forschung als Vektoren, um die Funktion von Genen zu untersuchen. Dazu wird ein Gen zunächst kloniert und im Labor in das Virusgenom eingebaut; danach werden Zellen infiziert, um die biologische Aktivität des Gens zu erkunden. Das Virus, das am häufigsten zu diesem Zweck benutzt wird, ist ein Mausvirus, das Murine Leukämievirus. Es ist auch vorgeschlagen worden, Retroviren in der Gentherapie einzusetzen, um eine gute Kopie eines Mutanten-Gens für Menschen mit Erbkrankheiten bereitzustellen. Mehr über den positiven Einsatz von Viren findet sich auf Seite 234.
Replikation von Retroviren
Retroviren gelangen in die Wirtszelle, indem sie ihre Membran mit derjenigen der Zelle verschmelzen und den inneren Core freisetzen, der zwei Kopien des Genoms enthält. Die einzelsträngige RNA wird durch das virale Enzym Reverse Transkriptase in doppelsträngige DNA umgewandelt. Die doppelsträngige DNA (rosa) wandert in den Zellkern und wird in das Wirtsgenom (blau) integriert. Dort werden die Messenger-RNAs und die neuen Genome genauso hergestellt wie die zellulären MessengerRNAs. Die Virusproteine werden im Cytoplasma synthetisiert und dienen dazu, neue Viruspartikel zusammenzubauen.

Bindung
Fusion und Eindringen
RNA-Spleißen
Wenn DNA in RNA übersetzt wird, enthält sie Teile, die für die Translation in Proteine benutzt werden (Exons), und Teile, die nicht dazu benutzt werden (Introns). Diese Introns tragen andere Informationen, die damit zusammenhängen, wie und wann ein Gen aktiviert wird. Sie müssen aus der RNA entfernt werden, bevor diese als reifer mRNA-Strang eingesetzt werden kann, ein Prozess, der von Ribonucleoproteinen durchgeführt wird, die teils aus kleinen snRNA-Molekülen, teils aus Proteinen bestehen. Die meisten eukaryotischen Gene haben Introns; einige Viren benutzen diese Strategie ebenfalls.

Zusammenbau

Knospung


Reifung Nucleus
DonorstelleVerzweigungsstelleAkzeptorstelle Intron Intron
Ribonucleoproteine (snRNPs) Spliceosom
(nur Exons)
Viren der
Das Tollwutvirus tritt in Nordamerika und Europa nur selten auf, findet sich aber häufiger in Teilen der Welt, in denen Heimtiere nicht geimpft werden. Das Virus wird durch Bisse übertragen, und die Krankheit, die es auslöst, führt zu geistiger Verwirrung und Tod.
Beim Menschen findet die initiale Infektion mit dem Tollwutvirus oft Monate vor dem Auftreten der ersten Krankheitssymptome statt, sodass es sehr schwierig ist, die Quelle festzustellen. Zu den ersten Symptomen gehören in der Regel Fieber und Kopfschmerzen, doch sie schreiten zu einer Gehirnentzündung fort. Die Krankheit ist grundsätzlich immer tödlich. Es gibt einen dokumentierten Fall eines jungen Mädchens in Wisconsin, USA, das 2003 eine Tollwuterkrankung nach intensiver Behandlung – einschließlich eines induzierten Komas – überlebte, heute als MilwaukeeProtokoll bekannt. Aber auch wenn es inzwischen einige Berichte über weitere Fälle gibt, denen zufolge Menschen, bei denen dieses Protokoll angewandt wurde, überlebt haben, sind diese Fälle nicht gut dokumentiert, und im Allgemeinen ist dieses Protokoll als ineffektiv aufgegeben worden. Die Impfstoffe gegen Tollwut sind sehr wirksam, und in vielen Teilen der Welt werden die meisten Heimtiere geimpft. Menschen, die ihre Arbeit in Kontakt mit tollwütigen Tieren bringen kann, wie Veterinär:innen und Feldforscher:innen, können sich ebenfalls impfen lassen. Die Erkrankung schreitet anfangs so langsam fort, dass Menschen auch noch nach einem erfolgten Kontakt erfolgreich geimpft werden können.
GRUPPE
FAMILIE
GATTUNG
GENOM
VIRUSPARTIKEL
WIRTE
KRANKHEIT/SYMPTOME
ÜBERTRAGUNG IMPFSTOFF
V Rabdoviridae
Lyssavirus lineare, einzelsträngige RNA mit etwa 11000 Nucleotiden, die für 5 Proteine codiert, welche anschließend in funktionale Einheiten gespalten werden behüllte, projektilförmige Teilchen, rund 180 nm lang und 75 nm breit
Säuger, experimentell auch in Vögeln und Reptilien
Tollwut
Bisswunden inaktiviertes Virus
Früher bestand die einzige Behandlungsmöglichkeit nach einem Kontakt in der Verwendung von Immunsera (die aus Tieren wie Pferden und Schafen gewonnen wurden) und erforderte eine Reihe schmerzhafter Injektionen. Manchmal wird diese Behandlung noch zusammen mit dem Vakzin eingesetzt.
Das Hauptreservoir von Tollwutviren sind Wildtiere, einschließlich Fledermäusen, Waschbären, Stinktieren, wilden Hundeartigen (Füchsen) und verwilderten Haushunden. Vögel können ebenfalls infiziert sein, zeigen aber keinerlei Symptome. Humaninfektionen gehen häufig (aber nicht in Europa) auf Fledermäuse zurück, deren Bisse oft unbemerkt bleiben. Im Gegensatz zu vielen anderen Humanviren, die von Fledermäusen übertragen werden, erkranken auch Fledermäuse an Tollwut, doch gewöhnlich sterben sie nicht daran.
Kolorierte transmissionselektronenmikroskopische Abbildung eines mit dem Tollwutvirus (rot) infizierten Gewebeabschnitts, der zelluläre Einschlüsse (blau) aufweist.

AAbacavir 183
Acquired Immune Deficiency Syndrome (AIDS) 226
Adenoviren 33, 68, 71, 72, 73
Affenpockenvirus (Mpox-Virus) 73, 184
Afrikanisches Cassavamosaikvirus (ACMV) 267, 272–273
Afrikanisches Schweinepestvirus (ASFV) 276, 277
Ageratum-Yellow-Vein-Virus 213
Albatros, Laysan- 69
Algen,Viren von 52, 64
Algenblüte 203–204
Allele 158
Alphapapillomavirus 124, 125
Amerikanische (Ess-)Kastanie (Castanea dentata) 234, 235
Aminosäuren 12
Amphibien,Viren von 45
Amylase 222
Anpassung an verschiedene Umgebungen/Wirte 139, 140, 143, 152, 251
Antibiotika 182, 210
Antibiotikaresistenz 182, 236
Antikörper 163, 169, 170, 180, 190 therapeutische 182, 186 monoklonale 186
Aquakultur 41
Archaea, Archaeen (Reich) 30, 31, 36, 172
Aufbau der Zelle 108
Immunantwort 162, 172, 173
Argonautenprotein 171 Artgrenze, Überspringen der 148, 156, 214, 250, 268, 270 Arthropoden als Virusvektoren 117, 118, 119
Arzneimittel, antivirale 182–183
Ausbreitung von Viren siehe Übertragung
Ausrottung einer Krankheit 175 Autoimmunkrankheit 181
B
Baculoviren 44, 203, 218
Bakterien (Reich) 30, 31
Bakterien 139, 172
Aufbau der Zelle 10, 108
Immunantworten/
Verteidigungsstrategien 162, 172, 173, 192
Konkurrenten, KillerBakteriophagen 232, 233 Lyse durch marine Viren 196, 197, 198, 216
Bakterienviren siehe Bakteriophagen
Bakteriophage Q 158–159
Bakteriophagen 32, 33, 236
Einschleusen des Genoms in Bakterien 64, 111, 236 experimentelle Evolutionsstudien 142
Form/Aufbau 32, 33
Genomintegration 232, 233 im Boden 43
Killerphagen 232, 233
Lyse von Bakterien in Meeren 197, 198, 216
Lysogenie und lytische Infektionen 244, 245 mutualistische Beziehungen 227, 231
RNA, hochvariable Populationen 150
Therapie (bakterielle Infektionen) 236
Töten des Wirts, Plaque-Test 196, 236 und Gesundheit von Menschen/Tieren 227, 236
Baltimore, David 25
Baltimore-Klassifikation 14, 38–39
Banana-Bunchy-Top-Virus (BBTV) 274–275
Banana-Streak-Virus (BSV) 14, 42, 54–55
Bananen 54–55
Bananenblattlaus (Pentalonia nigronervosa) 274
Basen, in DNA und RNA 12, 66
Basenpaarung 12, 66, 67
Basstölpel 257
Bau von Viren 13, 16, 34–35 behüllte Viren 108, 109, 113
Beijerinck, Martinus 18, 20–21
Bell-Pepper-Alphaendornavirus (BPEV) 132–133
Benennung von Viren 14–15
Biodiversität 30–31, 36, 46
Biomasse sämtlicher Viren 36
Biotyp 212
Bitterorange, als Pfropfunterlage 265
Blattläuse 230, 238
Einfluss von Virusinfektion auf Flügelbildung 199, 230 Interaktionen Pflanzen-VirenBlattläuse 199, 230, 238 Übertragung von Pflanzenviren 117, 118, 119, 143, 147, 266, 274
Blumenkohlmosaikvirus 87 bodenbürtige Viren 43, 118
Bornaviren 205
Braune Zitrusblattlaus (Toxoptera citricida) 264, 265, 266
B-Zellen 163, 169, 170, 179
B-Gedächtniszellen 170, 178
C
Canines Morbillivirus 156
Canines Parvovirus (CPPV-2) 113–114, 268
Capsomer 32
Carnivore-Protoparvovirus 1 (CPPV-1) 268–269
Cassava/Maniok (Manihot esculenta) 272
Chenopodium quinoa 168
Chikungunyavirus (CHIKV) 130–131, 208
Cholera 227, 244
Chromosomen 36
Citrus, Gattung 265
Citrus-Tristeza-Virus 264–266
Codon 12
Concatemer 58, 70, 74
Coronaviren 8, 139 siehe auch SARS-CoV; SARSCoV-2
COVID-19 siehe SARS-CoV-2
CRESS-Viren (circular, Repencoding, single-stranded viruses) 50, 74
CRISPR 172, 173
Curvularia-Orthocurvulavirus 1 240–241
Curvularia-ThermotoleranceVirus (CThTV) 240–241
Cyanobakterien, marine 198, 216
Cytomegalovirus 37, 227, 242
d’Hérelle, Félix 192
Darmbakterien/Mikrobiom 166, 210, 227
Darwin, Charles 136, 137
Delbrück, Max 192
Deltavirus 46
Denguefieber (Knochenbrecherfieber) 190, 208
Denguevirus 179, 190, 191, 205
Densoviren 40, 41, 44
DICER 171
Diphtherie 227
DNA (virale) Integration ins Wirtsgenom 48, 111, 222, 224
Humanes Endogenes
Retrovirus K 56–57
Pararetroviren 86, 87
Retroviren 48, 84, 85, 87, 222, 224, 270
tiefe Evolution, virale «Fossilien» 151
DNA 10, 13
Sequenzierung 28, 29, 154
Struktur 10, 12, 13, 48, 66, 68
DNA-abhängige DNAPolymerase 50, 90, 154
DNA-abhängige RNAPolymerase 76
DNA-Genom 8, 10, 12, 13 virales siehe DNA-Viren
DNA-Ligase 68, 70
DNA-Polymerasen 50, 70, 73, 74, 90, 154
DNA-Viren 8, 13, 68–75, 154
dsDNA 13, 14, 36, 38–39, 65
ssDNA 13, 14, 36, 50, 74, 75
DNA-Viren-Replikation 50, 68–75
dsDNA-Viren 68–73, 90, 184
Nutzung von Wirtsenzymen 68, 73, 74
Rolling-Circle-Methode 50, 70, 74, 75
ssDNA-Viren 74–75, 92
Strand-Displacement-Methode 58, 72
Domänen des Lebens 30 Dörrobstmotte (Plodia interpunctella) 202
EEbolavirus 29, 46, 141, 152
Eindringen/Verlassen des Wirts 106–111
Durchbrechen der Zellwände 110–111
Membranfusion, behüllte Viren 108, 109
SARS-CoV-2 262, 263 unbehüllte Viren 108, 109 «Eine RNA, ein Protein»Strategie 76, 78, 79, 81
einzellige Organismen,Viren von 39, 52
Eiserne Lunge 206, 207
Elysia chlorotica (marine Nacktschnecke) 203
Emiliania huxleyi (marine Phytoplanktonart) 203–204
Endocytose 108
endogene Viren 48, 54, 56, 57, 222
Endornaviren 132
Entdeckung der Viren 28, 29
Enteroviren 78
Enterovirus C (Poliovirus) 78, 206, 210–211
Entzündung 166, 180
Epstein-Barr-Virus 88
Erbsenblattlaus (Acyrthosiphon pisum) 107, 231
Erkältung,Viren als Ursache 78, 128, 186
Erworbenes
Immunschwächesyndrom (AIDS) 226
Escherichia coli 192, 196, 227
Escherichia-Virus T7 (T7-Phage) 192–193
Eukaryoten (Domäne) 30, 31, 158
Viren von 38–39
Zellen von 10, 11, 13, 30
Evolution 136–139, 151, 201, 223 experimentelle 142–145
Evolution von Viren 138, 141, 151, 156, 214
Bakteriophagen 142
Geschwindigkeit 138–139, 142, 144, 178, 182
Rekombination und 144 von Varianten 148–150
Wirt-Virus-Interaktionen 147
Pflanzenviren 143
Exogenisierung 54
Exons 84, 85
F
fäkal-orale Viren, Übertragung 113, 114, 115, 116, 122
Felines Panleukopenievirus (FPLV) 268–269
Felines Parvovirus (FPV) 268–269
Feuerbrand 237
Fieber 166, 180
Fischviren 41
Fitness, biologische 140–141
Flaschenhals, Population 141, 145, 150
Flechten 222
Fledermäuse, als Virusvektoren 29, 46, 47, 98, 114, 261
Flug-Hafer (Avena fatua) 209
Formen von Viren 32, 33, 34–35, 37
«Fossilien»,Virus- 151
F-Pili 158
Franklin, Rosalind 18, 22–23
Frosch, Paul 18
Froschvirus 3 44
G
Gain-of-Function-Genetik 82
Galleria-mellonella-Densovirus 44
Gelbfieber 208
Gelbfiebermücke (Aedes aegypti) 130, 190
Gemeiner Samtfußrübling (Flammulina velutipes) 200, 201
Geminiviren 74, 152, 212, 272
Gendrift 256
Gene 13, 28, 32, 138
Übertragung/Transfer 96, 188, 197
genetischer Code 12, 18, 28–29
Genitalwarzen 124
Genom (viral) 8, 14, 32, 36, 38–39, 139
Baltimore-Klassifikation 14, 38–39
Größe 32, 50, 52–53
Komplementierung 150 Kopieren und Proofreading 67, 74, 76, 152
siehe auch DNA-Genom; RNA-Genom
Typen nach Wirtstypen 38–39
Genom, Definition und Aufbau 12
Genshift 256
Gentechnik 96, 188
Gentherapie 84, 237
Geschichte der Virologie 18 Glomerulonephritis 180 Grippe siehe Influenza
Größe von Viren 32, 33, 50, 52, 184
Große Wachsmotte (Galleria mellonella) 44
Gurkenmosaikvirus (CMV) 15, 49, 74, 78, 179, 214–215 experimentelle Evolution 143, 145 Impfung von Tabakpflanzen mit 179
Übertragung 118, 143
«gute» Viren 18, 222–247 Bekämpfung von Kastanienrindenkrebs 234 Einfluss auf Gesundheit von Mensch und Tier 226–227, 244 in Meeren 197, 198, 216 Kontrolle/Bekämpfung von Pathogenen 227, 234–237, 242
Schutz des Wirts vor Blattläusen 230, 238 Schutz des Wirts vor Stress 228–231, 240 Symbiogenese 222–224
H
Hämagglutinin (H), Influenzavirus 256, 257 hämorrhagisches Fieber 190
Hefen, «Killerviren» durch Toxin 232, 246
Helfervirus 49, 58, 246 helikale Viren 32, 33 Helikase 68, 70
Heparin 276
Hepatitis A 122, 182
Hepatitis-A-Virus (HAV) 114, 122–123
Hepatitis-B-Virus (HBV) 58, 180
Hepatitis-C-Virus (HCV) 122, 182
Hepatitis-D-Virus (HDV) 3, 58–59
Hepatitis-G-Virus (Pegavirus C) 226
Hepatovirus A (HAV) 114, 122–123
Herdenimmunität 255
Herpes-Simplex-Virus 1 (HSV-1) 154–155
Herpesviren 33, 71, 242
Histone 86
Hitzetoleranz bei Pflanzen 228, 240
horizontale Übertragung 112, 115, 203, 218, 274, 276 horizontaler Gentransfer 147
Hot Springs Panic Grass (Dicanthelium lanuginosum) 228, 240, 241
Hülle 32, 33, 64, 106, 108
Hüllprotein 32, 49
Humanes Adenovirus 3 71
Humanes Cytomegalovirus (HCMV) 37, 227, 242
Humanes Endogenes Retrovirus K (HERV-K) 56–57, 222
Humanes Endogenes Retrovirus W (HERV-W) 56–57
Humanes Herpesvirus 71
Humanes Herpesvirus 1 154–155
Humanes Immunschwächevirus (HIV) 182–183, 226, 270
Humanes Orthopneumovirus (RSV) 186–187
Humanes Papillomavirus 124–125
Humanes Respirator isches
Synzytial-Virus (RSV) 186–187
Hundestaupe 156
I
ikosaederförmige Viren 32, 33, 100, 101
Immungedächtnis 162, 170, 178
Immunität 162–165 nach Impfung 178–179
Immunreaktion/-antwort 162–163, 166–167, 178, 180–181, 186
adaptive/erworbene 172–173, 181 angeborene 166–168, 186 humorale 169, 170 Priming 168 überaktive 180
Unterdrückung durch parasitoide Wespen 224 Viren vermeiden/bekämpfen Abwehrreaktion des Wirts 148, 178, 186 Immunseren 182
Immunsystem 166–173, 180–181 adaptives (erworbenes) 162, 163, 169–173, 188 angeborenes 162, 163, 166–168, 180
Immuntoleranz 180–181 Impfung und Impfstoffe 174–179 Alphapapillomavirus 124 attenuierte VirusLebendimpfstoffe 177, 210
DNA-Impfstoffe 190 Pocken 175, 184
Impfstoffe auf RNA-Basis 178 Influenza (Grippe) 179 Polio (Kinderlähmung) 177, 210
Rinderpest 156
SARS-CoV-2 (COVID-19) 178, 179 Tollwut 98, 175, 176 T-Zell-Impfstoffe 179 Virus-Totimpfstoffe 176 von Pflanzen 179 Indiana-Vesiculovirus 152–153 indigene Bevölkerung 209, 232 Infektionen bakterielle (Mensch) 236 latente 242 Infektionszyklus/prozess 64–67, 108, 109
SARS-CoV-2 262, 263 siehe auch Eindringen/Verlassen des Wirts; Replikation von Viren
Verpackung des viralen Genoms 74, 80, 81 Influenza (Grippe) 252–258 bei Vögeln 257, 258 Impfstoffe 179 Pandemien 250, 252–255, 256, 257, 261
Influenzavirus/-viren 33, 37, 256, 257 bei Vögeln 45, 256, 258 bei Meeressäugern 40, 41 Genetik und Stämme 178, 256, 257
Genomstruktur, Replikation 78, 79, 256
Immunreaktion auf 178, 257 Segmente 78, 79, 256, 257 Übertragung und Ausbreitung 116, 252–253, 254 ursprünglicher und Zwischenwirt 258 Varianten 178, 256, 257
Insekten als Überträger von Pflanzenviren 117, 118, 147, 199
Pflanze–Insekt–VirusInteraktionen 199, 230, 238
Populationsregulation durch Viren 44, 202–204, 218
Stechmücken 29, 90, 107, 117, 120, 130, 205
Verhalten, Einfluss von Viren auf 199, 205, 218, 230, 238
Virusübertragung auf Tiere 29, 90, 107, 117, 118
Insektenviren 44, 94, 108 Pflanzen als Vektoren 118 Übertragung 118, 203, 218 Interferone 167, 180, 242
International Committee on Taxonomy of Viruses (ICTV; Internationales Komitee für die Taxonomie von Viren) 14, 15, 36
Introns 84, 85 invasive Arten 209 Iridoviren 16, 205
Iwanowski, Dmitri 18
J Jenner, Edward 175, 184
K
Kältestress 228
Kaninchen, Populationskontrolle 209
Kastanienr indenkrebs 234, 235 Katzenseuche, Katzenstaupe 268, 269
Killer-Bakteriophage (Killerphage) 232, 233 «Killerviren», bei Hefe 232 Kinderlähmung siehe Poliomyelitis
Klassifikation von Viren 14–15, 30, 38–39
Kleine Braune Fledermaus (Myotis lucifugus) 46 Klimawandel 266
Klonierung von Viren 210
Kohlenstoffkreislauf, globaler 197, 198, 203, 204
Krebs 46, 88, 237 mit Viren assoziiert 56, 87, 100
Kryo-EM (Kryo-Elektronenmikroskopie) 16
kryptische Viren 238
Kuhpocken, Kuhpockenvirus 75, 184
L
La-Crosse-Virus (LACV) 205 Läsionen, nekrotische 96, 168 Lebendimpfstoffe, attenuierte 177, 210
Loeffler, Friedrich 18
Lymantria-dispar-MultipleNucleopolyhedrosis-Virus (LdMNPV) 218–219
Lyse 196, 197, 198, 216 Lysogenie 244
M
Maize-Streak-Virus 267
Mallon, Mary 164, 165 Marek-Krankheit 65 marine Viren 36, 40–41, 52, 196–198, 216 Masern 156 Masernvirus 156 Masken, medizinische 116, 128, 262
Maus-Herpesvirus 226, 227
Meer,Viren im 36, 40–41, 52, 196–198, 216
Meeressäugerviren 40, 41 Mehlige Apfelblattlaus (Dysaphis plantaginea) 230
Menschen und Viren 14, 46 Messenger-RNA (mRNA) 64, 65, 68, 74, 76
Bildung durch DNA-Viren 65, 74
Bildung durch Pararetroviren 87
Bildung durch Retroviren 84, 85, 86
Bildung durch RNA-Viren 76, 78, 79, 80, 81, 82, 96
Caps und Poly-A-Schwanz 76, 77
Meyer-Zitrone 264, 265
Middle East Respiratory Syndrome (MERS) 29, 46, 148, 261
Mikrobiota/Mikrobiom 166, 210, 227
«Mikrochromosom» 86
Mitochondrien 10, 11, 30, 201, 222
Monokultur 146, 147, 266
Morbilliviren 156–157
Morus bassanus 257
Mottenschildlaus/«Weiße Fliege» (Bemisia tabaci) 212, 272
Mouse-Gammaherpesvirus-68 (MHV-68) 242–243
Murid-Gammaherpesvirus 4 (MHV) 242
Murines Leukämievirus 84
Mutationen 67, 76, 136, 139 «neutral» oder positive/negativ 136
Anhäufung während der Replikation 136, 138, 139, 154
Keimbahn 136
Nucleotid-Analoga und 182 RNA-Viren 67, 76, 136, 138, 139, 152 und Komplementierung 150
Mutualismus/mutualistische Beziehung 212, 222, 224, 230
Bakteriophagen 227, 231
Blattläuse, mit Viren 230, 231
fakultativ, bei Pflanzen-VirusSymbiose 229
Pflanzen und Pilze, Hitzetoleranz 228, 240
Myxomavirus und Myxomatose 209
Pathogen-assoziierte molekulare Muster (PAMPs oder MAMPs) 167
N
Nanoviren 274
Narnaviridae 201
Nematoden, als Vektoren 118
Neuraminidase (N), Influenzavirus 256, 257
Niesen 113
Norovirus 227
Nucleotid-Analoga 182
Nucleotide 12, 13, 66, 67
Nucleus (Zellkern) 68, 73
Nutz-/Kulturpflanzen
«Spillover» von Viren aus Wildpflanzen auf 43, 250 Beziehung von Pilzen,Viren und 200, 201
Infektion mit Geminiviren 272
Pandemien 264, 265, 266 und Viren 43, 208, 251 Virusresistenz 164
O
Okazaki-Fragmente 70, 71
Ökosysteme, Funktion von Viren in 196–219
Onkogene 87, 88, 100
Orfvirus 33
Organellen 10, 30
P
Pandemie 250, 251 COVID-19 siehe COVID-19 (Pandemie)
Influenza siehe Influenza (Grippe)
Pflanzenvirus 264–266
Pandoravirus dulce 52
Pandoravirus salinus 52–53
Panicum-Mosaic-Virus (PMV) 118, 126–127
Paprika 132–133, 238
Paramecium 108
Paramyxovirus 33
Pararetroviren, Replikation 86–88, 102
Parvoviren 74
Pasteur, Louis 176
Pathogene (virale) 250–277 Kampf zwischen Virus und Wirt 180–181, 183 Rolle von Bakteriophagen 227, 236
Viren zur Kontrolle von 234–237, 242
Pegavirus C (Hepatitis-G-Virus) 226
Penton-Partikel 71
Pepper-Cryptic-Virus 1 (PCV-1)
238–239
Pest, Beulenpest 226, 227, 242 Pflanzen
Abwehrmoleküle, Schäden durch 180–181
adaptive Immunreaktion 170, 171, 188 als Vektor für Insektenviren 118 angeborene Immunreaktion 162, 167, 168
Bau der Zelle 10, 11, 108
Bekämpfung von Pathogenen durch Phagen 236
Beziehungen zu Pilzen 200, 201, 228, 240
Gentechnik, Experimente 96, 188
Impfung 179
Insekt-Virus-PflanzeInteraktionen 118, 199, 230, 238
nekrotische Läsionen 96, 168 systemisch erworbene Resistenz 168
Pflanzenviren 38–39, 43, 200–201
Bedeutung 264 bei Nutzpflanzen 43, 208, 251 Benennung 14
Eintritt/Austritt und Bewegung in Pflanzen 110, 111
Epidemien 208, 214, 264–266
Genomtypen 36 globale Ausbreitung 208 Hitzebehandlung 212 Infektion von Insekten 94, 108, 110, 199 Infektion von Pilzen 200 Pandemien 264–266 Pflanze-Insekt-VirusInteraktionen 199, 230, 238
Replikation 88, 94 Resistenz gegen 164
RNA-Silencing und 178, 180 Schutz von Pflanzen vor Blattläusen 230, 238
Schutz von Pflanzen vor Stress 228, 229
Überspringen der Artgrenze 250, 251
Übertragung 110, 112, 117, 118, 126, 132, 147, 199 Untersuchung der Evolution 143
Vektoren siehe Vektoren
Phage siehe Bakteriophage
Phagentherapie 236 phiX174 ( X174) 33
Phoebastria immutabilis 69 Phylogenetik 142
Phytoplankton 198, 203–204 Picornavirus 33 Pilze 108, 200
Besiedlung von Wildpflanzen 200, 228, 240 Beziehungen zu Pflanzen 200, 201, 228, 240
Immunreaktionen bei 162, 170, 171 Infektion durch Pflanzenviren 214
Virusübertragung bei 111
Pilzviren 14–15, 43, 200–201, 228 Hitzetoleranz bei Pflanzen und 228, 240
Überleben der Edelkastanie und 234
Übertragung 111, 112 Pithovirus sibericum 33, 52
Plaque-Test 196, 236 Plasmamembran 108 Plasmodesmen 110, 111 Plazenta, Entwicklung 222, 223
Pocken, Echte 174, 175, 209, 232
Ausrottungsprogramm 175, 184
Impfung 175, 184
Poliomyelitis («Kinderlähmung», Polio) 78, 206–207, 210
Impfstoff 177, 210 Poliovirus (Enterovirus C) 78, 206, 210–211 Polydnaviren 224
Polymerase-Kettenreaktion (polymerase chain reaction, PCR) 102 Polyomavirus 33
Polyprotein 78, 79, 84, 188
Population, Flaschenhals 141, 145, 150
Populationsregulation durch Viren 202–204, 218
Porcines Circovirus 1 (PCV-1) 33, 50–51
Porcines Circovirus 2 (PCV-2) 50
Potato-Spindle-Tuber-Viroid (PSTVd) 49, 60–61
Poxviren 68, 73
Prägenom 80, 81, 87
Präimmunisierung 126
Primer 68, 74, 75
Prokaryoten 10, 31
Zellen von 10
Proofreading, Kopieren des Genoms 67, 74, 76, 152
Protease 84, 188
Hemmer 182
Proteine 12, 13, 32
Synthese 12, 13, 76, 78, 79
Protisten 30, 31, 38–39, 52, 113
Immunantworten 162, 170, 171
Provirus 56
Quasispecies 158
Qubevirus durum (Bakteriophage Q ) 158, 159
Quinoa 168
Ranavirus 45
Raspberry-Latent-Virus 81
Reassortment (Neuverteilung) 256
genetisches 144
Reed,Walter 18
Reich (Taxonomie) 30
Reis,Virenbefall 94, 95, 102, 132, 265
Rekombination 144, 154
Reoviren 94
Replikation von Viren 15, 50, 68–88
Anhäufung von Mutationen 138
DNA-Viren siehe DNA-VirenReplikation
Pararetroviren 86–88, 102 Retroviren 64, 84–85, 86, 100
RNA-Viren siehe RNAViren-Replikation
Replikation von Viroiden 89
Rep-Protein 50, 74, 75
Resistenz 164, 168
Restriktions-Modifikationssystem (R-M) 192
Retroviren 48, 100, 222 als Genvektoren 84
DNA-Integration ins Wirtsgenom 56, 57, 84, 85, 87, 222, 224, 270
endogene (humane) 48, 56, 57, 222 im Vogelpockenvirus 90
Infektionsprozess 84, 85, 86
Replikation 64, 84–85, 86, 100
Symbiogenese 222
Reverse Genetik 82
Reverse Transkriptase 25, 38–39, 100
Hemmer 182
reverse Transkription 14, 38–39
Rhabdoviren 33, 79
Rhinoviren 78, 178
Rhinovirus C 128–129
Ribozyme 58, 60, 89
Rice-Ragged-Stunt-Virus (RRSV) 94, 95
Rice-Tungro-Bacilliform-Virus (RTBV) 102, 103
Rice-Tungro-Spherical-Virus 102
Rinderpest 156
RNA Pol II 89
RNA 13, 18
Aufbau 13, 66, 76, 77
Impfstoffe auf RNA-Basis 178, 179
Satelliten- 49, 246
Spleißen 84, 85 subgenomische 78, 79, 96
RNA-Genom siehe RNA-Viren
RNA-Interferenz 170, 171, 178, 180
RNA-Polymerase 65, 76, 201 RNA-abhängige 76, 80, 132, 152, 158
RNA-Primer 68
RNAs, small interfering (siRNAs, kleine Interferenz-RNAs) 170, 171
RNA-Silencing 162, 170, 171, 178, 180, 188
RNA-Viren 8, 13, 14
angeborene Immunität gegen 186
Anzahl der Gene 68
dsRNA 14, 64, 80–81, 94, 178, 246
hochvariable Populationen 150
mRNA-Bildung,Translation 76, 78, 79, 80, 81, 82, 96
Mutationen 67, 76, 136, 138, 139, 152
natürliche Selektion 138
Partikelgröße und Anzahl 81
Pathogen-assoziierte molekulare Muster (PAMPs) 167
Proteinsynthesestrategien 79 Quasispecies 158
Rekombination 144
Retroviren 14, 38–39, 48
ssRNA(-) 13, 14, 38–39, 58, 82, 152
ssRNA(+) 13, 14, 38–39, 48, 82
Vermeiden der WirtsImmunantwort 178, 246
RNA-Viren-Replikation 76–88, 171
dsRNA-Viren 64, 80–81, 94, 178, 246 in Viroplasmen 78, 80, 83 intermediäre dsRNA 83
Retroviren 14, 64, 84–85, 86, 100
ssRNA(-)-Viren 78, 79, 83
ssRNA(+)-Viren 82, 96
Verpackung des Genoms 80, 81
Rolling-Circle-Replikation 50, 58, 60, 74, 75, 89
Rolling-Hairpin-Replikation 74, 75
Röntgenkristallografie 16, 18, 22
Rous, Peyton 100
Rous-Sarkom-Virus (RSV) 100–101
S
Saccharomyces-cerevisiae-VirusL-A (ScV-L-A) 15, 246–247
SARS-CoV (Schweres-AkutesRespiratorisches-SyndromCoronavirus) 259, 260
SARS-CoV-2 18, 28–29, 261–263
Impfstoff auf RNA-Basis 178, 179
Krankheitsmechanismus 262, 263
Mutationen 67, 262
Pandemie 18, 210, 261–263
Spike-Protein 67, 261–262, 263
Übertragung 106, 116
Ursprung (möglicher) 46, 261
Wellen und Namen der Varianten 262
Satelliten-RNA 49, 246
Satellitenviren 49, 58, 59, 126
Schädlingsbekämpfung, biologische 218
Scharka-Virus (Plum-Pox-Virus) 79, 251, 267
Schwammspinner (Lymantria dispar) 202, 218
Schweinepest, Afrikanische (ASF) 276
Schweres Akutes Respiratorisches Syndrom (SARS) 259–263
Schweres-AkutesRespiratorisches-SyndromCoronavirus (SARS-CoV) 259, 260
Schweres-AkutesRespiratorisches-SyndromCoronavirus-2 siehe SARSCoV-2
Simianes Immunschwächevirus (SIV) 205, 270–271
Selektion, natürliche 136, 138, 142 small interfering RNAs (siRNAs, kleine Interferenz-RNAs) 170, 171
Sojabohnen 146
Spanische Grippe 252–255
Spleißen (Splicing) 84, 85
St. Augustine Decline 118, 126
Stabilität von Viren 113 Stammbaum des Lebens 151 Stechmücken,Virusübertragung durch 29, 90, 107, 117, 121, 130, 205
Strand-Displacement-Replication 68, 72
Stress,Viren schützen Wirte vor 228–231, 240 subgenomische RNA 78, 79, 96
subvirale Einheiten 49, 89
Suszeptibilität für Infektionen 165 Symbiogenese 222–225
Symbionten,Viren als 222 Symbiose 222–225, 229
Syncytin 56, 223
Synechococcus-Virus Syn5 (SVSyn5) 216, 217
TT4-Bakteriophage 111
T7-Phage 192, 193
Tabak-Etch-Virus (TEV) 188–189
Tabakmosaikvirus (TMV) 8, 15, 18, 19, 32, 78, 96–97, 113, 143
Temlin, Howard Martin 23
Temperaturtoleranz 166, 228, 240 terrestrische Viren 42–47
T-even-Coliphage 33
T-Gedächtniszellen 170
Thripse 199
Tiere,Wirbeltiere 30, 45 adaptive Immunreaktion 162, 163, 169–170 angeborene Immunreaktion 162, 163, 166–167 Bau der Zelle 11, 108 Tierreich 30
Tierviren 38–39, 46
Stechmücken als Vektoren 29, 90, 107, 117, 130, 205
Übertragungswege 114, 115, 117
Toleranz, gegenüber Infektionen 164, 165
Tollwut 98, 176, 182
Impfstoff 98, 175, 176
Tollwutvirus 37, 98–99, 114, 205
Tomate (Solanum lycopersicum) 49, 60, 208, 212
Tomato-Spotted-Wilt-Virus 76
Tomato-Yellow-Leaf-Curl-Virus 43, 212–213
Topoisomerase 68, 70
Torque-Teno-Virus 1 (TTV1) 92, 93
Transfer-RNA (tRNA) 76–77
transgene Pflanzen 188
Transkription 13, 14
Translation 13, 80, 81
Transmission siehe Übertragung
Trinkwasser 210
Trockenstress 228, 229
Tubulovirus 33
Tulpen und Tulip-Breaking-Virus (TBV) 16, 17
Tungro-Krankheit 102
T-Zellen 163, 169, 179, 180, 190
T-Zell-Impfstoffe 179
U
Übertragung (Transmission) von Viren 29, 46, 54, 90, 113–116 durch Blattläuse siehe Blattläuse durch Lebensmittel/Wasser 113, 114, 115, 116, 122 durch Luft 114, 115, 116, 128 durch Tröpfchen 115, 116 durch Vektoren siehe Vektoren Flaschenhals 145 horizontal 112, 115, 203, 218, 274, 276 in Körperflüssigkeiten 114, 115, 116
Insektenviren 118, 203, 218
Pflanzenviren 110, 112, 117, 118, 126, 132, 147, 199 sexuell 114, 124 vertikal 112, 132, 203, 218, 238 Wirt-zu-Wirt-Kontakt 113–114, 115, 116
Übertragung von Viroiden 49 unbehüllte Viren 108, 109 «Uncoating» von Viren 64, 90
V
Vacciniavirus 33, 175, 184–185 «Vakzinverabreichungssystem», VSV als 152
Varianten, Evolution von 148–150, 154, 158 Variolation 174
Variolavirus 37
Vektoren 107, 117–119, 276 Blattläuse siehe Blattläuse Mottenschildlaus/«Weiße Fliege» 212, 272
Stechmücken 29, 90, 107, 117, 120, 130, 205
Verhalten des Wirts, Einfluss von Viren auf 199, 205, 218, 230, 238
Vesicular-Stomatitis-Virus (VSV) 152–153
Vesikel (Bläschen) 109
Vibrio cholerae 227, 244
Vibrio-Virus CTXphi (CTX ) 244–245
«Viral Shunt» des Meeres 197, 198
Viren, die Archaeen infizieren 32, 38–39, 111
Virenarten/Virentypen 36
Viroide 49, 60, 89
Virologie 18–19, 28–29
Virom 42 des Menschen 227
Viroplasma 78, 80, 82
Virulenz 165
Virushülle 108, 109, 113
Viruspopulationen 148, 149, 150
Vögel,Viren 45, 90, 256, 258
Vogelgrippe 257, 258
Vogelpockenvirus 69, 90–91
W
Warzen 124 wasserlebende Viren 113, 114, 115
«Weiße Fliege» 212, 272
Wespen 205, 224, 225, 231
Westliche Honigbiene (Apis mellifera),Viren der 44
West-Nil-Virus 45, 107
Wheat-Streak-Mosaic-Virus 267
Wirbellose Tiere, Immunreaktionen 162, 170, 171
Wirt/Wirte
Enzyme 67, 68, 73, 74, 158
Evolution, Einfluss von Viren 147, 148
Integration der viralen DNA siehe DNA (virale)
Integration Immunreaktion siehe Immunreaktion; Immunsystem; Immunität Schutz vor Stress 228–231, 240 veränderte Wirtsökologie 206–208
Verhalten siehe Verhalten verschiedene
Viruspopulationen in 148, 149
Verteidigung, physische Barrieren 106, 166
Virus-Fähigkeit zur Infektion verschiedener 94, 108, 152, 199, 200, 214 Wirtsspektrum von Viren 147 Wirt-zu-Wirt-Kontakt 113–114
Y Yellowstone-Nationalpark 228, 240
Z
Zellen, Bau 10, 11, 108 Zellmembran 108 Zellwände 10, 106, 108 Durchbruch von Viren 110–111
Zikavirus 9, 179, 190 Zwischenwirte 46








Sie sind mikroskopisch klein, vermehren sich rasend schnell, sind überaus anpassungsfähig und bemerkenswert effizient. Viren kommen praktisch überall vor, bewegen sich in uns und um uns herum und haben die Macht, innerhalb kürzester Zeit ganze Systeme lahmzulegen. Doch Viren sind keineswegs nur unheilbringende Krankheitserreger, im Gegenteil: Sie können sogar dazu beitragen, Krankheiten zu heilen, sei dies bei Pflanzen, Tieren oder bei uns Menschen.

In diesem reich bebilderten Buch nimmt Sie Marilyn Roossinck mit in die vielfältige Welt der Viren. Sie zeigt auf, was Viren sind und woher sie kommen, wie sie sich verbreiten und welch großen Einfluss sie auf uns und unsere Umwelt, auf Tiere, Pflanzen und das Gleichgewicht der Ökosysteme, haben –im negativen, aber auch im positiven Sinn. Sie sind mikroskopis nzen aben –n Sinn. mehren sich rase un

ISBN 978-3-258-08407-7
