
7 minute read
Predicción de posibles biomarcadores microbianos autóctonos en Penaeus vannamei, identificados mediante metaanálisis y modelización metabólica a escala genómica
Los datos de la secuencia de amplicones 16S y el modelado metabólico, basado en el genoma, se combinaron para la aplicación en acuicultura, con el propósito de encontrar biomarcadores nativos que puedan emplearse mejor, para desarrollar formulaciones probióticas, que aprovechen los microorganismos beneficiosos para el cultivo de Penaeus vannamei, al tiempo que limitan el crecimiento de patógenos.
Por: Redacción de PAM*
Con el advenimiento de la secuenciación de alto rendimiento, los conjuntos de datos del metagenoma se han vuelto cada vez más accesibles. El enfoque metagenómico independiente del cultivo ha facilitado un análisis exhaustivo de los datos del microbioma con fines preventivos y de diagnóstico. Incluso, en el sector de la acuicultura, los consorcios microbianos son cruciales para mejorar la sostenibilidad y la productividad de los organismos acuáticos. Diversos factores amenazan la sostenibilidad y el rendimiento de las especies acuícolas, uno de los cuales se relaciona con la infección causada por microorganismos patógenos. Por lo tanto, comprender los consorcios microbianos esenciales y significativos simplificaría la predicción de la incidencia de enfermedades.
En organismos acuáticos, el enfoque basado en la secuencia de amplicones se ha utilizado para identificar microbiomas, su composición y funciones. El estudio actual se centra en Penaeus vannamei, cuyo cultivo ha aumentado rápidamente para satisfacer la creciente demanda de proteínas comestibles de alta calidad. A pesar de esto, el cultivo de P. vannamei se ve constantemente obstaculizado por factores abióticos y bióticos. Uno de los desafíos se relaciona con las enfer- medades emergentes causadas por bacterias, virus y hongos, que afectan la sostenibilidad del camarón. Es por ello que, este artículo presenta el resumen de un metaanálisis realizado con la finalidad de lograr una comprensión más sólida y consistente de los estados de salud frente a los estados de enfermedad en P. vannamei
Métodos
Los datos de la secuencia de amplicón utilizados. para el metaanálisis. se recuperaron de la base de datos NCBI SRA (descargada en septiembre de 2021). Los estudios recolectados se relacionaron con especies de P. vannamei asociadas con una enfermedad, considerando solo el tejido del huésped o muestras de intestino. Inicialmente, la búsqueda produjo 13 estudios con 838 conjuntos de datos. Sin embargo, se descartaron algunos estudios debido a la falta de información sobre el hábitat.
El conjunto de datos de la enfermedad incluido en este metaanálisis corresponde a eventos de enfermedad de heces blancas (DMA, por sus siglas en inglés), virus del síndrome de la mancha blanca (WSSV) y enfermedad necrótica hepatopancreática aguda (AHPND).
Los estudios disponibles contrastaron en las subregiones del gen 16S rRNA, como V3-V4, V4, V1-V2, V2 mix y V3 mix secuenciados con diferentes plataformas de secuenciación. Debido al número limitado de estudios con metadatos adecuados, no fue posible implementar una tecnología de secuenciación uniforme o una región hipervariable común. Finalmente, se consideraron seis estudios con un total de 259 conjuntos de datos.
Resultados
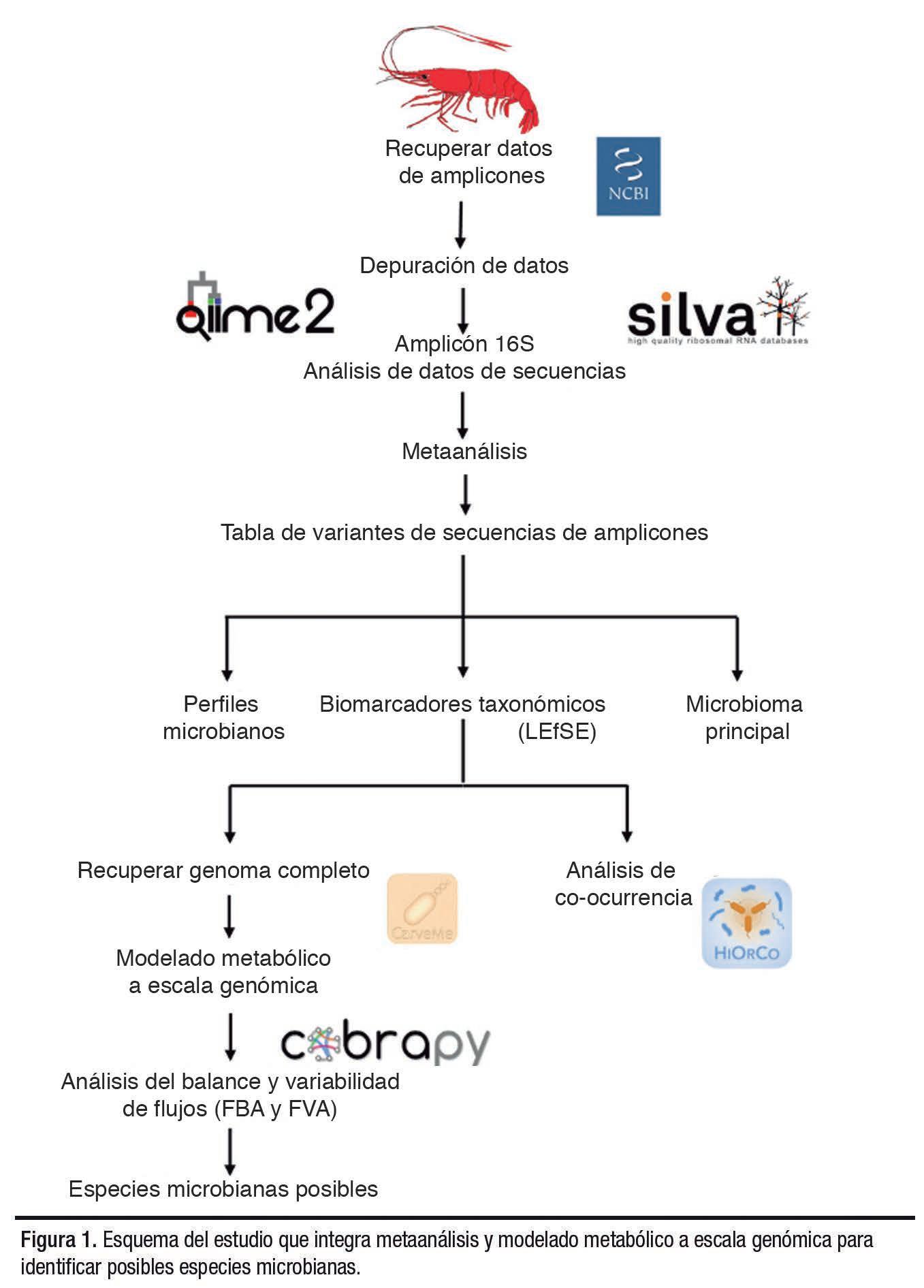
Los seis estudios incluyeron datos que abarcaban 259 conjuntos de datos (117 sanos y 142 enfermedades). Los criterios de inclusión para este metaanálisis fueron estudios basados en la secuencia de amplicones sobre P. vannamei en estados sanos y de enfermedad. Para identificar un biomarcador taxonómico potencial, un solo estudio puede no ser suficiente; por lo tanto, se realizó un metaanálisis que podría servir como un biomarcador representativo y biológicamente significativo. Además, se llevó a cabo un enfoque de modelado metabólico, basado en restricciones, para capturar las capacidades metabólicas de los biomarcadores taxonómicos en diferentes entornos nutricionales e inferir las interacciones entre especies. La Figura 1 muestra el flujo de trabajo que describe los pasos clave seguidos en el estudio.
Abundancia microbiana a nivel de filo y género en estados saludable y de enfermedad
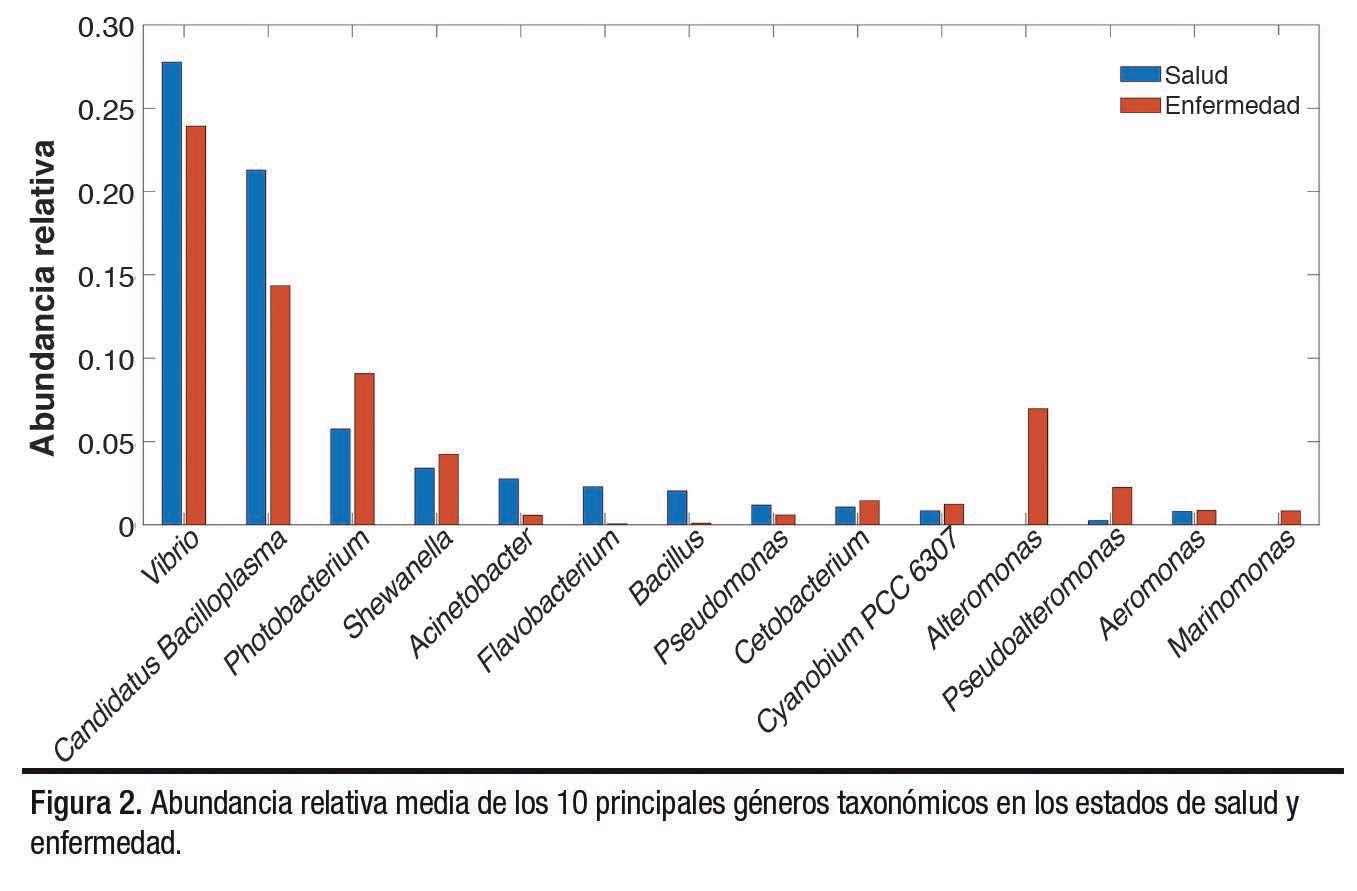
Se examinaron los cinco principales filos y géneros dominantes, calculando la abundancia relativa media en conjuntos de datos sanos y de enfermedades. Un filo o género con una abundancia media de ≥ 0.01 se consideró abundante. A nivel de filo, Proteobacteria (62% en estado sano vs. 72% en estado de enfermedad) y Firmicutes (30% en sano vs. 20% en estado de enfermedad) son los principales representantes, seguidos por Bacteroidota, Actinobacteriota y Cyanobacteria en ambos estados según la abundancia relativa media. Cuatro géneros dominaron tanto los estados sanos como los de enfermedad en orden descendente de magnitud, a saber: Vibrio, Candidatus Bacilloplasma, Photobacterium y Shewanella. Sin embargo, la abundancia relativa media de estos géne- ros fue marginalmente diferente en ambos estados, como se muestra en la Figura 2. Curiosamente, el género Alteromonas se presentó solo en el estado de enfermedad, y el género Acinetobacter fue más abundante en el estado saludable, mostrando una menor abundancia en el estado de enfermedad.
El cultivo de P. vannamei se ve constantemente obstaculizado por factores abióticos y bióticos. Uno de los desafíos se relaciona con las enfermedades emergentes causadas por bacterias, virus y hongos que afectan la sostenibilidad del camarón.
Detección de biomarcadores microbianos en estados saludable y de enfermedad
El análisis discriminante lineal tamaño del efecto (LEfSe, por sus siglas en inglés), de biomarcadores taxonómicos, se llevó a cabo en el conjunto de datos combinado para estimar si había una diferencia significativa en la abundancia relativa entre el estado saludable/enfermedad. LEfSe identificó 32 géneros beneficiosos y 73 géneros de enfermedades como biomarcadores prospectivos con un tamaño de efecto mayor de dos y un valor de p < 0.05. Los cinco principales biomarcadores sanos pertenecían al filo Firmicutes y Proteobacterium, incluidos los géneros Candidatus, Bacilloplasma, Acinetobacter, Exiguobacterium, Lactobacillus y Shimia. Por otro lado, los cinco principales biomarcadores de enfermedades pertenecían al filo Proteobacteria, incluidos los géneros Alteromonas, Photobacterium, Pseudoalteromonas, Halomonas y Marinomonas. La abundancia relativa media correspondiente a los biomarcadores taxonómicos dominantes en el estado saludable y de enfermedad se representa en la Figura 3. Además, fue interesante observar que la mayoría de los biomarcadores de enfermedades identificados pertenecían al filo Proteobacteria.
Discusión
Las comunidades microbianas naturalmente existentes realizan distintas tareas vitales, como la degradación de la materia orgánica, el reciclaje de los nutrientes y el control del desarrollo de infecciones microbianas (Bentzon-Tilia et al., 2016). Es fundamental comprender el estado de salud y las comunidades micro- bianas asociadas a la enfermedad, para mantener la salud del huésped. Numerosas investigaciones basadas en la secuencia de amplicones 16S han intentado desentrañar el papel de los microbios, pero los hallazgos extraídos de un estudio pueden no descifrar el espectro completo de microbios responsables de los estados de salud/enfermedad. Por lo tanto, estudiar múltiples estudios en un metaanálisis tiene el potencial de hacer inferencias sobre las características comunes asociadas con la salud del huésped en diferentes hábitats. vannamei y determinan las interacciones entre especies exhibidas por los biomarcadores microbianos en diferentes entornos de nutrientes. Como un aporte al manejo sostenible de la enfermedad, el objetivo de este estudio es identificar especies autóctonas beneficiosas en P. vannamei esenciales para mantener un buen estado saludable y controlar el crecimiento de especies patógenas.
Aunque, se reportaron varios trabajos sobre la perspectiva de la salud humana, muy pocos estudios se realizan sobre especies acuáticas, pero no se extendieron para comprender las interacciones metabólicas entre ellas (Cornejo-Granados et al., 2018; Yu et al., 2018). La identificación de biomarcadores taxonómicos en la acuicultura no es nueva; sin embargo, la mayoría de las investigaciones se centran en biomarcadores taxonómicos basados en un solo estudio, lo que implica la necesidad de un análisis mucho más completo.
En este contexto, el presente metaanálisis identificó los biomarcadores taxonómicos centrales que impulsan las diferencias en los estados de salud/enfermedad en P.
El estudio identificó Acinetobacter como uno de los géneros prevalentes y dominantes en el estado saludable. Acinetobacter fue previamente detectado y reportado en camarones sanos para ayudar a suprimir bacterias dañinas (Farzanfar et al., 2006). Se informó que los principales géneros revelados en el estado saludable de este estudio fueron Acinetobacter y Candidatus bacilloplasma, los cuales actúan a modo de eje central que conecta la importante población bacteriana en la red del hepatopáncreas de camarón sano (Wang et al., 2020). Además, los géneros Exiguobacterium en estado saludable han mejorado el crecimiento y la supervivencia en P. vannamei y han proporcionado ventajas probióticas (Cong et al., 2017; de Mello Júnior et al., 2021). Los miembros de Lactobacillus identificados en este estudio son probióticos conocidos y han sido ampliamente estudiados por sus habilidades para mantener la salud animal. Shimia sp, otro género identificado en estado saludable en este análisis, se ha informado que produce metabolitos beneficiosos y degrada las toxinas (Duan et al., 2019).
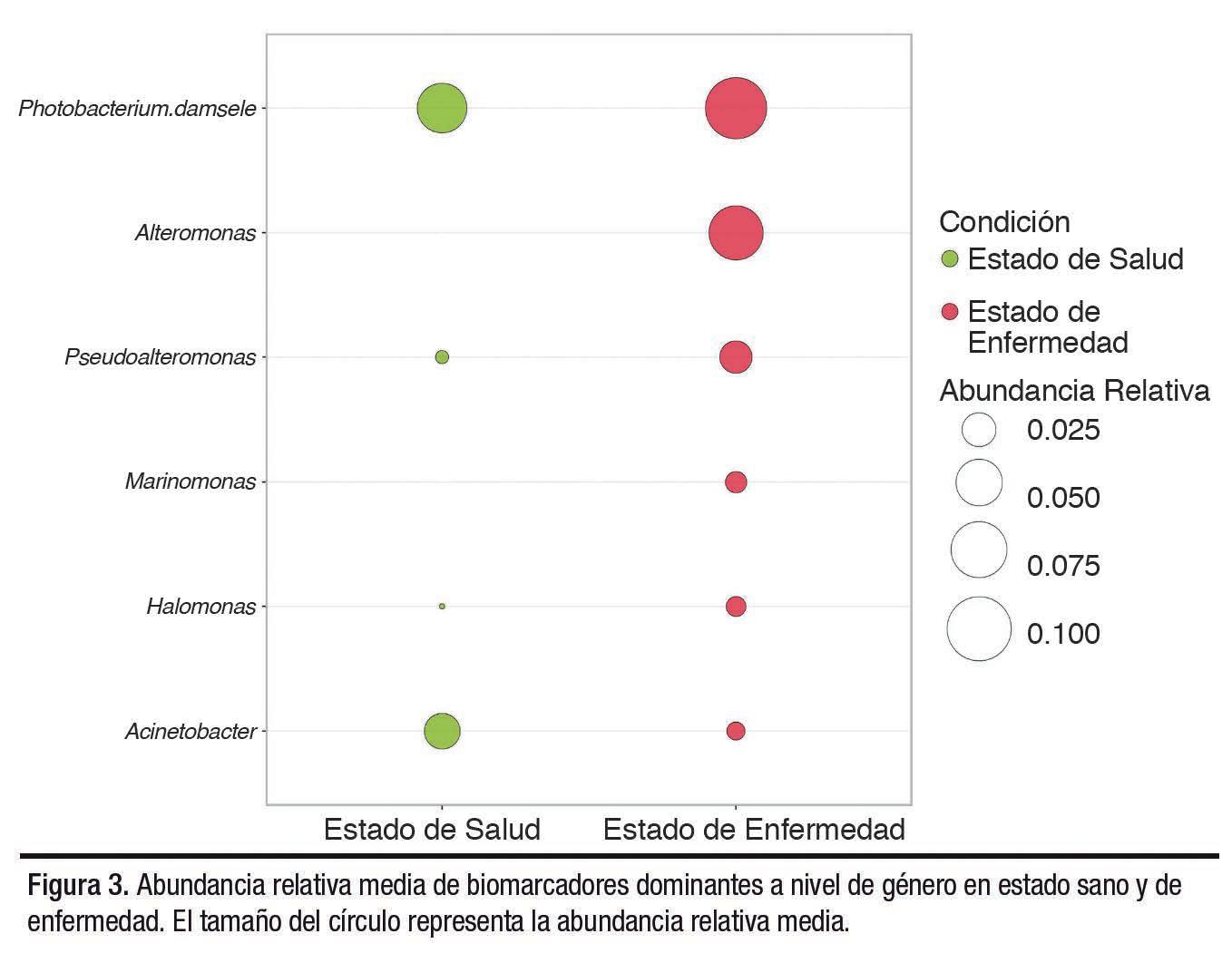

A pesar de que se encontró Vibrio, en ambos estados, apoyando estudios previos (de Souza Valente et al., 2021; He et al., 2020), se considera como bacteria oportunista y causa enfermedades cuando el organismo se encuentra bajo estrés. En general, los géneros sanos reportados en el presente estudio son comparables con estudios anteriores, lo que confirma la exactitud de este enfoque.
Está bien establecido que los ácidos orgánicos se emplean en la conservación de alimentos, como aditivos para alimentos y para controlar patógenos (Ng et al., 2017). Aunque muchas especies beneficiosas o probióticas se usan en la acuicultura, la falta de consistencia y rendimiento en diferentes condiciones es una preocupación importante. Dado que las interacciones de las especies microbianas varían en diferentes entornos de nutrientes, es crucial identificar el entorno favorable que controla el crecimiento de las especies de enfermedades. Como resultado, estos hallazgos agregan valor al demostrar que la sacarosa facilita las interacciones parasitarias (es decir, limita el crecimiento de la enfermedad), lo que es consistente con investigaciones anteriores que destacaron el papel de la sacarosa y las especies benefi- ciosas en la mejora de la calidad de agua, el crecimiento de P. vannamei y la composición microbiana.
Conclusión
Por primera vez, los datos de la secuencia de amplicones 16S y el modelado metabólico basado en el genoma se combinaron para la aplicación en acuicultura, con el propósito de encontrar biomarcadores nativos que puedan emplearse mejor para desarrollar formulaciones probióticas que aprovechen los microorganismos benéficos para el cultivo de P. vannamei, al tiempo que limitan el crecimiento de patógenos. El análisis basado en secuencias de amplicones combinado con el modelado metabólico proporcionó información sobre las interacciones metabólicas y el impacto de los entornos de nutrientes y, finalmente, permitió seleccionar posibles especies benéficas para acelerar los experimentos. Se empleó esta estrategia de combinación para la aplicación de probióticos en acuicultura, la cual se aplicó previamente a la microbiota intestinal humana. Los géneros clave identificados en este estudio podrán usarse para preparar una formulación y reponer los consorcios microbianos sanos. Con el fin de fomentar el cultivo saludable de camarones, estos géneros esenciales pueden reducir la gravedad de las enfermedades eliminando bacterias patógenas oportunistas y potenciando las asociaciones benéficas internas.
La versión informativa del artículo original está patrocinada por: GRUPO GAM.
Esta es una versión resumida desarrollada por el equipo editorial de Panorama Acuícola Magazine del artículo “IN SILICO PREDICTION OF POTENTIAL INDIGENOUS MICROBIAL BIOMARKERS IN PENAEUS VANNAMEI IDENTIFED THROUGH META-ANALYSIS AND GENOME-SCALE METABOLIC MODELLING”escritoporNEELAKANTANTHULASIDEVIKA,VINAYA KUMAR KATNENI,ASHOK KUMAR JANGAM, PANJAN NATHAMUNI SUGANYA,MUDAGANDUR SHASHI SHEKHAR AND KARINGALAKKANDY POOCHIRIAN JITHENDRAN - Central Institute ofBrackishwaterAquaculture,Chennai, India. La versión original, incluyendo tablas y figuras, fue publicada en ENERO de 2023 en ENVIRONMENTAL MICROBIOME. Se puede acceder a la versión completa a través de https:// doi.org/10.1186/s40793-022-00458-6










