7 minute read
8.5 DETERMINAZIONE DELLA CONTAMINAZIONE VIRALE NEI CAMPIONI DI SUPERFICIE O ARIA
from La sanificazione degli ambienti sanitari: standard tecnici, monitoraggio, gestione rischio clinico
by edicomsrl
Come precedentemente accennato, essendo i virus sono parassiti intracellulari obbligati, a differenza dei microrganismi a crescita autonoma (batteri, miceti), il loro isolamento è attuabile unicamente mediante l’uso di colture di cellule bersaglio (permissive) in vitro, in grado di sostenere la replicazione dei virus ricercati.
Questi sistemi di indagine biologica, molto utili nella ricerca e nello sviluppo di terapie e vaccini, sono però di difficile realizzazione in laboratori che hanno come scopo il monitoraggio microbico ambientale, richiedendo tempi di indagine prolungati e la disponibilità di laboratori con livelli di isolamento idonei alla classe di rischio manipolata, strumentazioni costose e elevata esperienza del personale tecnico.
Le acquisizioni in biologia molecolare hanno invece reso possibile l’identificazione rapida di numerosi virus senza necessità di isolamento biologico. Le metodiche molecolari, molto utilizzate per la diagnosi rapida, possono quindi essere applicate con successo anche nel monitoraggio ambientale, contribuendo significativamente alla comprensione della trasmissione/diffusione dei virus e al loro controllo (si pensi a quanto utilizzato nel controllo della pandemia da SARS-CoV-2).
I tempi di realizzazione di tali analisi (con risultati ottenibili entro 24 h dal momento del campionamento) possono consentire l’adozione immediata di misure di contenimento in grado di minimizzare la trasmissione ad altri pazienti o personale sanitario, prevenendo outbreaks di infezione a tutto l’ospedale, alla comunità e a livello globale.
Estrazione del materiale genetico dal campione raccolto: Le metodiche molecolari si avvalgono di procedure di evidenziazione del materiale genetico del/dei virus ricercati, che deve quindi essere estratte dal materiale in esame.
L’estrazione può essere fatta manualmente o automaticamente: esistono a questo scopo numerosi kit commerciali che consentono di estrarre DNA o RNA con o senza l’ausilio di estrattori automatici o semi-automatici. Alcuni kit consentono l’estrazione simultanea di entrambi i tipi di acidi nucleici e sono da preferire soprattutto quando il materiale a disposizione per l’analisi è scarso, in quanto consentono di evidenziare sia virus a DNA che virus a RNA nello stesso campione.
I kit commerciali sono forniti di manuali d’uso del fornitore: tali istruzioni devono essere seguite nella realizzazione del protocollo di estrazione, in quanto sono diversificati a seconda della ditta produttrice.
Il materiale estratto, solitamente eluito in soluzione sterile a bassa forza ionica priva di RNasi e DNasi (anch’essa solitamente fornita con il kit di estrazione), deve essere preferibilmente controllato e quantificato mediante lettura spettrofotometrica alla lunghezza d’onda di 260/280 nm. In caso di campioni molto scarsi, è possibile effettuare le successive analisi downstream evitando tale passaggio e utilizzando tutto il campione a disposizione senza quantificarlo.
Analisi molecolare:
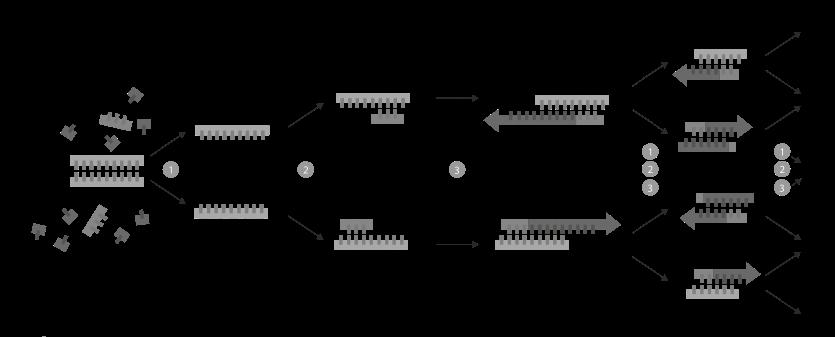
Il materiale genetico estratto viene analizzato mediante amplificazione di uno o più geni virali bersaglio attraverso la Polymerase Chain Reaction (PCR, reazione di polimerizzazione a catena), già ampiamente nota e utilizzata soprattutto a scopo diagnostico in virologia.
La PCR è in grado di analizzare soltanto sequenze di DNA, pertanto se si vogliono includere nell’analisi anche virus con genoma a RNA è necessario retrotrascrivere a DNA le sequenze di RNA estratte dal campione: la retrotrascrizione viene effettuate mediante l’enzima retrotranscrittasi (RT, reverse transcriptase), di derivazione virale (Retrovirus), in grado di sintetizzare DNA a partire da uno stampo di RNA. Una volta ottenuto il DNA complementare, è possibile effettuare la PCR su questa molecola. La procedura prende in questo caso il nome di RT-PCR, cioè PCR dopo Retro Trascrizione. L’analisi mediante PCR si basa sulla amplificazione delle sequenze bersaglio cercate in modo tale da renderle evidenti: se nel campione sono presenti le sequenze ricercate (anche in piccolissima quantità) la amplificazione molecolare ne consentirà l’evidenziazione; viceversa, se nel campione non sono presenti le sequenze bersaglio, l’amplificazione non genererà alcun segnale ed il campione risulterà negativo.
Per effettuare tale analisi è necessario uno strumento apposito detto termociclatore, in grado di realizzare tutte le fasi termiche previste dalla amplificazione mediante PCR (Figura 5).
I risultati possono essere quantitativi, in caso si possa effettui una curva standard di amplificazione utilizzando il bersaglio in quantità note e scalari, oppure qualitativi, in caso ci si riferisca più semplicemente ad una soglia di positività: al di sopra della soglia i campioni sono considerati positivi, al di sotto della soglia sono considerati negativi. La soglia è stabilita dal fornitore del sistema di analisi, e si basa su prove ripetute e standardizzate effettuate prima della commercializzazione del sistema. La soglia esprime il numero di cicli necessari per avere la comparsa di un segnale di amplificazione della sequenza: più il numero di cicli è basso, maggiore sarà il quantitativo di sequenza bersaglio presente nel campione; al contrario, un numero di cicli alto esprime assenza o scarsità della sequenza bersaglio. Di norma, la soglia di positività è posta tra 35 e 40 cicli di amplificazione (cicli soglia, CT). Attualmente la maggior parte dei kit diagnostici per SARS-CoV-2 si basano su questo tipo di analisi (Figura 8).
Le reazioni di PCR e RT-PCR, sia qualitative che real-time quantitative, possono essere interamente disegnate e messe a punto in-house, ma comportano competenze professionali nella messa a punto e validazione del sistema di analisi. In commercio esistono invece numerosi kit già validati (molti già CE-IVD, utilizzabili anche per diagnostica) per la rilevazione di molteplici patogeni virali, che possono essere utilmente adoperati senza necessità di messa a punto e particolare esperienza in biologia molecolare.
Al momento, le PCR quantitative real-time rappresentano il sistema preferito di analisi, per la loro versatilità, rapidità e capacità di fornire direttamente il risultato SI/ NO senza necessità di ulteriori passaggi.
8.6 INDICATORI DI PROCESSO
Per la peculiarità delle procedure usate nelle indagini molecolari è indispensabile inserire indicatori di efficienza delle procedure utilizzate.
In particolare è indicato includere nelle analisi:
1. un controllo dell’efficienza di estrazione degli acidi nucleici: ad esempio sequenze sintetiche di acido nucleico analogo a quello virale (DNA o RNA) a titolo noto; la presenza e la concentrazione di tali molecole dovrà essere evidenziata dall’analisi mediante PCR, RT-PCR, qPCR o RT-qPCR.
2. un controllo dell’efficienza di retrotrascrizione (in caso di virus con genoma a RNA): ad esempio sequenze di RNA a titolo noto (possono essere le stesse utilizzate per il controllo di efficienza dell’estrazione); la presenza e la concentrazione di tali molecole dovrà essere evidenziata dall’analisi mediante RT-PCR o RT-qPCR.
3. un controllo dell’efficienza di amplificazione: ad esempio sequenze di DNA a titolo noto che contengano la sequenza bersaglio; la presenza e la concentrazione di tali molecole dovrà essere evidenziata dall’analisi mediante PCR, RT-PCR, qPCR o RT-qPCR (controlli positivi di amplificazione; i controlli positivi diluiti scalarmente potranno essere inoltre usati per costruire una curva standard utile alla quantificazione del bersaglio, ottenuta per interpolazione)
4. controlli negativi di estrazione e di amplificazione (bianchi): data l’elevata sensibilità delle metodiche che impiegano l’amplificazione degli acidi nucleici, è essenziale includere controlli negativi dei processi di estrazione e analisi, per evitare il rischio di falsi positivi
8.7 INDICATORI DI RISULTATO MICROBIOLOGICO PER LA RICERCA VIRALE
Al momento non esistono parametri di riferimento quantitativi che definiscano il carico virale eventualmente “tollerabile” in quanto non associato ad effettivo rischio infettivo, e non si hanno notizie circa la soglia quantitativa di rischio per la diffusione del contagio.
E’ noto che la patogenicità e la capacità di causare infezione nell’ospite dipendono dalla carica virale, ma è altresì noto che la carica virale necessaria a provocare un’infezione varia a seconda dello stato di salute del soggetto.
Poichè la suscettibilità del paziente ospedalizzato è particolarmente elevata nei confronti di qualsiasi tipo di infezione (sia essa di origine batterica, micotica o virale), nella valutazione di indicatori microbiologici della contaminazione virale appare utile ricercare virus patogeni e riferirsi a risposte del tipo ON/OFF.
Non essendo presenti nei virus sequenze di tipo conservato che possano universalmente essere utilizzate per rilevare più tipologie di virus contemporaneamente, ogni analisi sarà mirata ad evidenziare la presenza di uno o più virus associati a specifiche patologie, mediante reazioni di PCR singole (singleplex) oppure mediante pannelli di reazioni in grado di evidenziare simultaneamente più tipi di sequenze (multiplex).
E’ consigliabile utilizzare sistemi validati e certificati CE-IVD, che garantiscono la validità del risultato ottenuto e minimizzano il rischio di falsi negativi e/o positivi.
E’ inoltre consigliabile includere nell’analisi controlli di processo e di amplificazione, ove non già presenti e previsti dai sistemi commerciali.
In un’ottica più generale, oltre alla ricerca di specifici virus patogeni, potrebbe essere utile anche la ricerca di virus diffusi nell’ambiente anche da soggetti sani (come ad esempio il TTV), che possono contribuire ad evidenziare la efficacia delle azioni di sanificazione. Ciò non costituisce comunque prova di rischio biologico.
Tuttavia, anche in considerazione delle risorse da dedicare all’analisi della contaminazione virale, appare più utile ricercare la presenza di virus che siano specificamente correlati a patologie che possono insorgere nel paziente ospedalizzato in seguito a contatto diretto e/o indiretto con tali agenti, piuttosto che la ricerca generalizzata di numerose tipologie di virus.
Tra i virus noti per essere presenti in ambiente ospedaliero, si propongono i seguenti gruppi come riferimento per la valutazione della contaminazione virale ospedaliera:
- Contaminazione da aerosol: coronavirus, virus influenzali, adenovirus
- Contaminazione da feci: norovirus, rotavirus
- Contaminazione da sangue: virus dell’epatite B
Inoltre, visti i recenti dati di letteratura, si ritiene potenzialmente utile anche il monitoraggio di specifici virus responsabili di outbreaks, come ad esempio il SARSCoV-2, o di virus che possano rappresentare un utile parametro di valutazione della contaminazione virale nell’ambiente considerato, come il Torque Teno Virus (TTV).
Per quanto riguarda virus specifici, sono già disponibili kit diagnostici CE-IVD utilizzabili per la ricerca anche ambientale di numerosi virus: SARS-CoV-2, pannelli di virus respiratori, pannelli di virus enterici, ecc. La ricerca ambientale di questi virus è tuttavia caratterizzata dalla mancanza di riferimenti procedurali univoci, in quanto al momento mancano procedure standard specificamente indirizzati al monitoraggio ambientale, così come valori soglia di riferimento per la valutazione del rischio infettivo. Tuttavia, sono in corso di stesura procedure standard di riferimento almeno per alcuni tipi di virus (come ad esempio il SARS-CoV-2 per Accredia), e si auspica un rilascio di tali documenti nell’immediato futuro, in modo che tutte le procedure di indagine possano essere standardizzate ed uniformate. In caso di virus specificamente legati ad outbreaks, anche una risposta qualitativa SI/NO potrebbe essere indicata, in quanto tali virus dovrebbero essere assenti o presenti soltanto in tracce per poter considerare un ambiente privo di rischio infettivo specifico.
La contaminazione ambientale da TTV potrebbe essere invece utilizzata come parametro generale di rischio di contaminazione ambientale da virus. Infatti, il TTV è un virus nudo con genoma a DNA a singola elica, appartenente alla famiglia Anelloviridae, che infetta la maggior parte del genere umano e viene di continuo rilasciato dai soggetti infetti e dai portatori asintomatici [Schmitz et al., 2019]. Il virus non è letao a manifestazioni patologiche specifiche nell’uomo, ma il suo rilascio aumenta nei pazienti ospedalizzati e nei soggetti immunosoppressi (Honorato et al. 2021; Lapa et al. 2021), il che lo rende un possibile marker di contaminazione virale di origine antropica, per monitorare e controllare il potenziale rischio infettivo legato alla contaminazione da virus nell’ambiente di ricovero. Essendo un virus sprovvisto di inviluppo pericapsidico e dotato di genoma a DNA, risulta inoltre particolarmente resistente nell’ambiente esterno, e quindi più facilmente isolabile e identificabile, rispetto a virus dotati di inviluppo e genoma a RNA, molto meno resistenti. A tale scopo, esistono attualmente procedure e kit che consentono di rilevare e quantificare il TTV (Kulifaj et al. 2018), che potrebbero essere standardizzate per l’applicazione alla rilevazione ambientale, in modo da poter fornire anche dei parametri soglia per il rischio infettivo, analogamente a quanto ottenuto per batteri e miceti.




