
9 minute read
Detección del Virus iridiscente decápodo 1 con sonda TaqMan con PCR en tiempo real
Autores: Liang Qiu a Xing Chen a Xiao-Meng Guo a,b Wen Gao a,c
Ruo-Heng Zhao a Qing-Li Zhang a Bing Yang a Jie Huang a,b,c, *
a Laboratorio de Ciencia de la Pesca Marina y Procesos de Producción de Alimentos, Laboratorio Piloto Nacional de Ciencia y Tecnología Marina (Qingdao), Laboratorio de Control de Enfermedades de Organismos para Maricultura, Ministerio de Agricultura y Asuntos Rurales, Laboratorio de Qingdao Epidemiologia y Bioseguridad para la Maricultura, Instituto de Investigación Pesquera Yellow Sea, Academia China de Ciencias Pesqueras, Qingdao 266071, China b Escuela de Ciencias e Ingeniería Marina, Universidad Agrícola de Qingdao, Qingdao 266109, China c UShanghai Ocean University, Shanghai 201306, China
huangjie@ysfri.ac.cn
Una enfermedad emergente ha causado una alta mortalidad y graves pérdidas de camarón, langostino y cangrejo de río cultivados en China. El agente etiológico de la nueva enfermedad es un virus de ADN bicatenario llamado Virus iridiscente decápodo 1 (DIV1) (Chen et al., 2019a; Qiu et al., 2017; Qiu et al., 2019a; Xu et al., 2016). El DIV1 fue descrito por primera vez por Xu et al. (2016) como Cherax quadricarinatus iridovirus (CQIV) y por Qiu et al. (2017) como Virus iridiscente de hemocitos de camarón (SHIV). Los signos clínicos incluyen atrofia del hepatopáncreas con pérdida de color, estómago e intestino vacíos, y un cuerpo de color rojizo (Qiu et al., 2017). El camarón Macrobrachium rosenbergii enfermo muestra un área blanca típica debajo del exoesqueleto en la base del rostrum llamado enfermedad de “cabeza blanca” o enfermedad de la “mancha blanca” (Qiu, et al., 2019a). Los individuos moribundos se hunden en el fondo de aguas profundas donde se acumulan los individuos muertos; alcanzando una mortalidad acumulada del 80%. El DIV1 ha sido reportado en algunas provincias costeras de China desde 2014, incluyendo las provincias de Zhejiang, Guangdong y Hebei (Qiu et al., 2017). Una vigilancia especializada en China en 2017–2018 reveló que el DIV1 se detectó en 11 de 16 provincias (Qiu et al., 2018c; Qiu et al., 2019b).
El DIV1 es un virus icosaédrico envuelto con un diámetro de aproximadamente 150 nm y un genoma de ADN bicatenario de aproximadamente 166 K pb (Li et al., 2017; Qiu et al., 2018b). Basado en dos aislamientos originales, SHIV 20141215 y CQIV CN01 (ICTV, 2019), el virus fue asignado por el Comité Internacional de Taxonomía de Virus (ICTV) como el único miembro del nuevo género Decapodiridovirus dentro de la Familia Iridoviridae. Las especies susceptibles actualmente conocidas incluyen C. quadricarinatus, Penaeus vannamei, Macrobrachium nipponense, M. rosenbergii, Procambarus clarkii y Exopalaemon carinicauda (Chen et al., 2019a; Qiu et al., 2017; Qiu et al., 2019a; Xu et al., 2016). Dos especies de cangrejo, Eriocheir sinensis y Pachygrapsus crassipes también se infectaron con DIV1 en la prueba experimental por inyección intramuscular (Pan et al., 2017). Vale la pena señalar que los resultados positivos han sido detectados en muestras de PCR de Penaeus chinensis, Penaeus japonicus, Macrobrachium superbum, Nereis succinea o algunos cladóceros (Qiu et al., 2017; Qiu et al., 2018c; Qiu et al., 2019a; Qiu et al., 2019b).
Entre los métodos de detección, la sonda TaqMan empleada en PCR en tiempo real es el método más sensible y específico para detectar y cuantificar virus de camarón. Algunos métodos de PCR en tiempo real pueden detectar una copia de ADN viral o ADNc para varios virus de camarón, como el Parvovirus hepatopancreático (VPH), baculovirus de Penaeus monodon (MBV) y virus de la mionecrosis infecciosa (IMNV) (Yan et al., 2009, 2010; Liu et al., 2013). La prueba de PCR en tiempo real con sonda TaqMan se estableció para detectar el gen ATPasa de DIV1 en nuestra investigación anterior (Qiu et al., 2018a). Este método fue específico, y el límite de detección fue de cuatro copias por reacción. Sin embargo, el fragmento amplificado usando este método se superpuso con el único método de PCR anidada publicado para DIV1 (Qiu et al., 2017), que podría deberse a la contaminación cruzada cuando ambos métodos fueron utilizados en laboratorio. Para evitar este riesgo, detallamos una nueva realidad del tiempo de ensayo de PCR para la detección de la proteína de la cápside principal (MCP) del DIV1.
Materiales y métodos Recolección de muestras y extracción de ADN El camarón P. vannamei se recolectó de granjas en Guangdong y Shandong, Provincia de China, y fueron almacenados a −80°C.
Se extrajo ADN de un total de 30 mg de tejido de cefalotórax de camarones utilizando un kit de ADN de animales marinos TIANamp (TIANGEN Biotech, Beijing, China), según las instrucciones del fabricante.
Primers y sonda Se diseñaron primers específicos y una sonda para el gen DIV1 MCP (Número de acceso de GenBank KY681039, MF599468, NC_040612) (Qiu et al., 2017; Qiu et al., 2018b; Li et al., 2017) usando AlleleID versión 5.01 (PREMIER Biosoft). La corriente arriba (142F 5′- AAT CCA TGC AAG GTT CCT CAG G -3’) y corriente abajo (142R 5′- CAA TCA ACA TGT Los cebadores CGC GGT GAA C -3’) produjo un amplicón de PCR de 142 pb. La sonda TaqMan (5’- CCA TAC GTG CTC GCT CGG CTT CGG -3’) fue sintetizada y marcada con 6-carboxifluoresceína (FAM) en el extremo 5’ y con N,N,N’,N′-tetrametil6-carboxirododamina (TAMRA) en el extremo 3’.
Ensayo de PCR en tiempo real y procedimiento Las reacciones de PCR en tiempo real se realizaron en un sistema de reacción de 20 μL que consiste en 10 μL 2 × Master Mix, 500 nM de cada primer (142F/142R), 200 nM de sonda TaqMan y 1 μL de plantilla de ADN usando FastStart Essential DNA Probes Master (Roche). Brevemente, después de una desnaturalización inicial a 95 °C durante 10 min, las amplificaciones se llevaron a cabo en 40 ciclos a una temperatura de fusión de 95 °C durante 10 y recocido a una temperatura de 60 °C durante 30 s. La amplificación, detección y análisis de data se realizó usando un instrumento de fluorescencia cuantitativa CFX-96 (Bio-Rad, EE. UU.).
Construcción de vectores de control positivo y estándares para la cuantificación El fragmento de ADN de 142pb se clonó en el vector pMD18-T (TaKaRa, Dalian, China). La secuencia del inserto fue confirmada por secuenciación (Sangon Biotech, Shanghai, China). La concentración del plásmido se determinó luego usando un NanoDrop 2000 (Thermo Fisher, América). Se estimó el número de copias del plásmido que contenía el 142bp, y se preparó una serie de diluciones como estándares.
Fig. 1. A: Prueba de especificidad analítica. Se extrajeron plantillas de ADN de camarón infectado con 1. DIV1, 2. WSSV, 3. IHHNV, 4. VAHPND, 5. EHP, 6. Camarones sanos; y 7. Agua (NTC). B: Prueba de sensibilidad analítica. 1–9: 1.2 × 10 8 a 1.2 copias μl -1 ADN de DIV1; 10: agua (NTC). C: Curva estándar.

Especificidad analítica y sensibilidad analítica Para probar la especificidad analítica, el ADN extraído de camarones infectados con el virus del síndrome de la mancha blanca (WSSV), necrosis infecciosa hipodérmica y virus de necrosis hematopoyética (IHHNV), Vibrio que causa la enfermedad de necrosis hepatopancreática aguda (VAHPND) y Enterocytozoon hepatopenaei (EHP), se utilizaron como plantillas en el ensayo de PCR en tiempo real. El control negativo, positivo y en blanco, fue el ADN de P. vannamei sano, el ADN de P. vannamei infectado con DIV1, y agua; respectivamente. Cada ensayo se realizó por triplicado. La sensibilidad analítica de la PCR en tiempo real se determinó utilizando una serie de diluciones de 10 veces del plásmido purificado pDIV1. La concentración del ADN plasmídico varió de 1.2 × 10 0 a 1.2 × 10 8 copias por reacción. Cada ensayo se realizó por triplicado.
Prueba de muestra clínica Se extrajo un total de 300 muestras de ADN del cefalotórax de P. vannamei con un estado de infección DIV1 desconocido. El ADN extraído fue probado por la PCR en tiempo real realizada en este estudio y anteriormente (Qiu et al., 2018a). Sensibilidad diagnóstica (DSe) y especificidad diagnóstica (DSp), se determinaron de acuerdo con el Manual de la Organización Mundial de Salud Animal (OIE) (OIE, 2019).
Resultados y discusión El gráfico de amplificación solo mostró el ADN extraído del cefalotórax de camarones infectados con DIV1 (Fig. 1A, curva 1); no se detectó el ADN a partir de muestras de camarones sanos o camarones infectados con WSSV, IHHNV, VAHPND y EHP (Fig. 1A, curvas 2–6). Por lo tanto, la prueba de especificidad analítica mostró que el ensayo de la PCR en tiempo real desarrollado en este estudio fue específico para DIV1.
La prueba de sensibilidad reveló que el ensayo de PCR en tiempo real podría detectar ADN de DIV1 tan bajo como 1.2 copias/reacción (Fig. 1B), más sensible que los métodos de PCR en tiempo real para DIV1 desarrollados previamente (Qiu et al., 2018a) o para WSSV (Durand y Lightner, 2002).
La curva estándar mostró un alto coeficiente de correlación (R 2 = 0.997) dentro del rango de 1.2 × 10 1 –1.2 × 10 8 copias de ADN/ reacción. La ecuación regresiva fue Ct = −3.169 · lg (copias de ADN de DIV1) + 42.524 (Fig. 1C). Por lo tanto, este nuevo ensayo de PCR en tiempo real podría usarse como una
herramienta cuantitativa apropiada dentro del rango de 1.2 × 10 1 –1.2 × 10 8 copias de ADN /reacción.
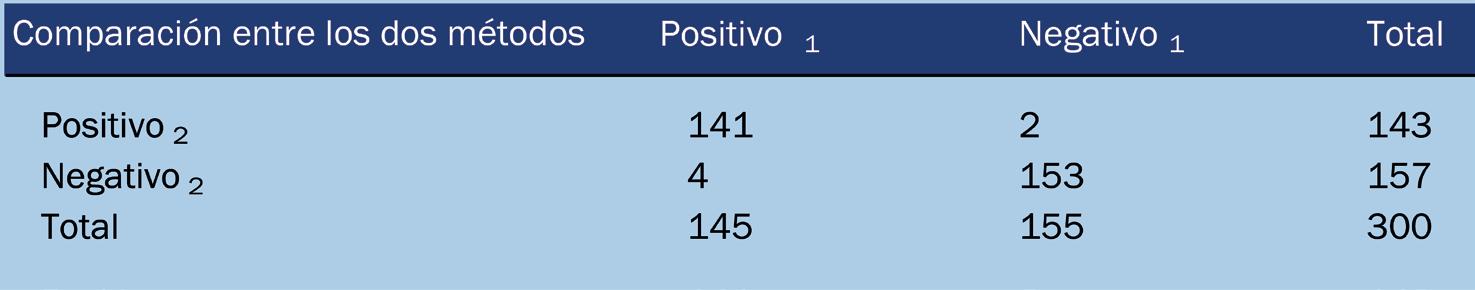
Los resultados de dos métodos de PCR en tiempo real para DIV1 acordaron que 141 muestras fueron positivas y 153 muestras negativas, y 6 muestras mostraron resultados diferentes para estos dos métodos de PCR en tiempo real (Tabla 1). En base a los resultados de las pruebas anteriores, el DSe (la proporción de muestras referenciales de animales infectados conocidos, fueron positivas en un ensayo) y el DSp (la proporción de muestras referenciales de animales no infectados conocidos, fueron negativos en un ensayo) del nuevo PCR en tiempo real fueron 97.2% y 98.7%, respectivamente.
En este estudio, desarrollamos una metodología TaqMan para DIV1 específica y altamente sensible basada en PCR en tiempo real. Debido a que la carga viral en larvas o juveniles puede ser muy baja, la sensibilidad del método de detección es crucial. Entre los métodos de detección publicados para DIV1, el nuevo de PCR en tiempo real es,
Tabla 1.- Resultados de detección de muestras con estado de infección desconocido por dos PCR en tiempo real.
Nota: Subíndice 1 = resultado probado usando PCR en tiempo real desarrollada por Qiu et al., 2018a; Subíndice 2 = resultado probado usando PCR en tiempo real desarrollada en este estudio.
por el momento, el más sensible (Qiu et al., 2017; Qiu et al., 2018a; Chen et al., 2019b). Además, evita el riesgo de contaminación cruzada cuando se utiliza el método de PCR anidado publicado, debido a que se orienta a diferentes genes.
Conflicto de interés y competencia Los autores declaran que no tienen ningún interés financiero o relación personal que podría haber influido el trabajo realizado en este estudio. Este trabajo fue apoyado por el Fondo Marine S&T de la Provincia Shandong para el Laboratorio Nacional Piloto de Ciencias del Mar y Tecnología (Qingdao) (2018SDKJ0502- 3); el Programa clave Nacional de I+D de China (2019YFD0900101); la Fundación de Ciencia Postdoctoral de China (2019M650170); el proyecto de innovación postdoctoral de la Provincia de Shandong (201902043); la Fundación de Investigación Postdoctoral Aplicada de Qingdao (2019); el Sistema de Investigación Agrícola de China (COCHES-47)•
Para más información sobre este artículo escriba a: huangjie@ysfri.ac.cn











